di Linda Celeste Montemiglio1,2, Adele Di Matteo1,2, Carmelinda Savino1,2, Barbara Illi1, Andrea Ilari1,2
1Istituto di Biologia e Patologia Molecolari, Consiglio Nazionale delle Ricerche (IBPM-CNR)
2Facility di Biocristallografia, Dipartimento di Scienze Biochimiche, Sapienza University of Rome
Indice dei contenuti
- Introduzione
- CoV-2: un altro virus con la corona
- La proteasi principale del CoV-2: l’origine dell’intero arsenale molecolare del virus
- La struttura cristallografica della proteina Mpro in complesso con l’inibitore N3
- Dalla struttura della Mpro del CoV-2 ai farmaci: il ruolo degli screening virtuali e high-throughput
- La proteina Spike del CoV-2: la chiave molecolare del virus
- Aspetti strutturali dell’interazione tra CoV-2-S e ACE2: la porta d’ingresso per l’invasione delle cellule umane
- Porte di accesso alternative: le integrine e il CD147
- L’RNA polimerasi RNA dipendente (RdRp) del CoV-2: la macchina di replicazione del virus
Approfondimenti: le tecniche
- Criomicroscopio a trasmissione elettronica: la macchina potente che fotografa le grandi biomolecole
- La cristallografia a raggi X
“Per i non esperti”
- Protein Data Bank (PDB)
- Proteasi
- Oligopeptide
- Struttura a barile β
- α-Elica
- IC
- Modello dell’interazione chiave-serratura
- Recettori
Bibliografia
Introduzione
La pandemia di COVID-19 attualmente in corso ha urgentemente spinto l’intera comunità scientifica a dedicare enormi sforzi, lavoro e risorse all’identificazione e allo sviluppo di nuove strategie farmacologiche per arrestare l’infezione da SARS-CoV-2 (di seguito indicato con CoV-2). Come nell’arte della guerra, per poter sconfiggere il nemico è fondamentale conoscere: com’è fatto il virus, qual è la sua forma? Come infetta le cellule umane? Come cresce, replica e si sviluppa nelle cellule ospite? Di che cosa ha bisogno per sopravvivere? Rispondere a tali domande fornisce le armi, le informazioni chiave a cui i laboratori di ricerca ambiscono per sviluppare vaccini e farmaci antivirali sicuri ed efficaci.
A molte di queste domande è già stato risposto. Ma come si possono ottenere queste informazioni senza poter vedere il nemico? Una particella virale e tutto il macchinario molecolare che usa per replicarsi e sopravvivere nelle cellule ospite non è né visibile a occhio nudo né usando un classico microscopio ottico. È qui che entra in gioco la biologia strutturale, il cui scopo è proprio quello di identificare la struttura tridimensionale delle macromolecole biologiche, come le proteine e gli acidi nucleici, e di correlarla con la loro funzione fisiopatologica. Questa disciplina scientifica si basa su tecniche estremamente avanzate che consentono di visualizzare e analizzare molecole invisibili e di combattere invisibili agenti patogeni. La conoscenza della forma, della struttura tridimensionale, e quindi della chimica del CoV-2 e di tutte le biomolecole responsabili dell’infezione e della replicazione virale costituisce il mezzo ottimale per sviluppare strategie di cura e per identificare, progettare e produrre molecole come vaccini e farmaci antivirali, che bersaglino e arrestino le sue funzioni e i suoi effetti dannosi sulla salute umana.
Al momento, le strutture di 13 delle 26 proteine di CoV-2 sono state depositate nella banca dati internazionale delle strutture di proteine (Protein Data Bank, PDB), ma ci si aspetta che il numero cresca rapidamente nel prossimo futuro.
Nei prossimi paragrafi, partendo dalla descrizione dell’architettura complessiva del virus, verranno descritte le strutture delle tre protein di CoV-2 che sono considerate attualmente come i migliori bersagli farmacologici potenziali, ovvero la proteasi principale Mpro, la proteina Spike (S) e l’RNA polimerasi dipendente dall’RNA (RNA dependent RNA polymerase, RdRp). Verrà anche analizzata la relazione che lega le strutture di queste molecule alle loro funzioni fisiopatologiche. Le tecniche di biologia strutturale citate sono descritte nella sezione Approfondimenti: le tecniche.
CoV-2: un altro coronavirus con la corona
Il primo isolamento documentato del CoV-2 a partire da campioni prelevati di pazienti infetti è stato realizzato all’ospedale Spallanzani di Roma1 e ha permesso di intraprendere lo studio del nuovo agente patogeno virale in diversi laboratori a livello internazionale. Le immagini delle particelle virali, isolate da persone di tutto il mondo, sono state ottenute usando un microscopio particolate che sfrutta elettroni anziché fotoni come sorgente di radiazione, ossia il microscopio elettronico a trasmissione (TEM). Le proprietà ottiche degli elettroni rendono possibile osservare oggetti fino a 1–10 000 volte più piccoli di 1 µm (10−6 m). Le immagini ottenute mediante TEM di CoV-2 hanno confermato che il virus appartiene alla famiglia dei coronavirus, data la tipica forma a corona della superficie esterna (Figura 1).
Ora sappiamo che il CoV-2 fa parte, più nello specifico, dei β-coronavirus, costituiti da un RNA a singolo filamento con senso positivo (v. Il mondo sorpredente del genoma di SARS-CoV-2), di circa 29,9 kilobasi (kb; 1 kilobase = 1000 basi). Una particella virale (il virione) di CoV-2 ha un nucleocapside composto dall’RNA genomico e ricoperto da proteine fosforilate che interagiscono con la membrana virale durante l’assemblaggio del virione, giocando un ruolo critico nel potenziare la replicazione del virus2. L’RNA genomico e il nucleocapside sono avvolti da un doppio strato di fosfolipidi in cui sono immerse diverse proteine che svolgono ruoli cruciali per l’infezione e la replicazione: la proteina S, la proteina di membrana (M), l’emoagglutinina esterasi (HE) e la proteina del rivestimento (E). La particella virale ha un diametro di 60–100 nm (10−9 m) e appare rotonda o ovale3.
Figura 1 La struttura di CoV-2. 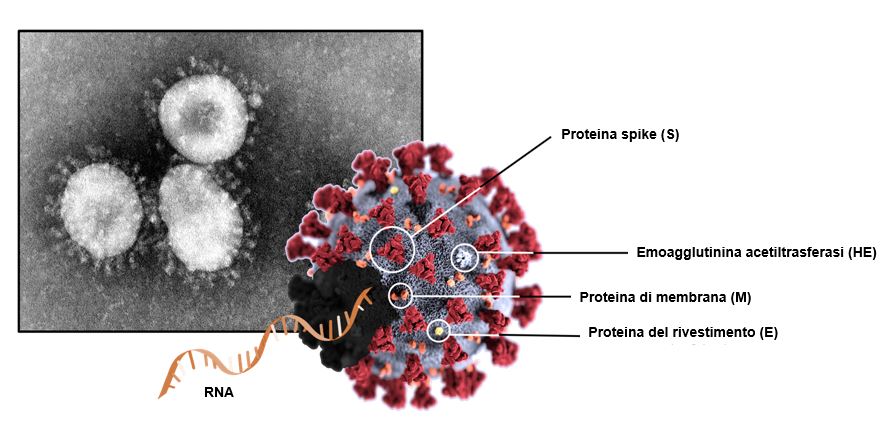
Immagine al microscopio elettronico a trasmissione di un coronavirus (fotografia in alto a sinistra). Le particelle virali appaiono bianche circondate da un fondo nero. Sono visibili le proteine Spike che “decorano” la superficie e che conferiscono al virione la caratteristica forma a corona. La ricostruzione grafica al centro dell’immagine mostra l’architettura molecolare del virus SARS-CoV-2. Il capside virale (in grigio), che contiene l’RNA a singolo filamento associato alle proteine del nucleocapside, è costituito da un doppio strato lipidico in cui sono inserite le proteine di superficie (S in rosso; E in giallo; M in arancio; HE in bianco). [Fotografia in alto a sinistra: CDC/Dr. Fred Murphy; Sylvia Whitfield; disegno in centro adattato da: CDC/Alissa Eckert, MSMI, Dan Higgins, MAMS]
La proteasi principale del CoV-2: l’origine dell’intero arsenale molecolare del virus
Durante l’infezione della cellula ospite, il genoma virale agisce come RNA messaggero (v. Il mondo sorpredente del genoma di SARS-CoV-2) e dirige la sintesi di due grandi poliproteine (pp1a e pp1ab) che contengono al loro interno proteine più piccole necessarie alla produzione di nuove particelle virali all’interno delle cellule infette. Tale insieme di proteine comprende: un complesso di replicazione/trascrizione, diverse proteine strutturali necessarie a costruire virioni e due proteasi4,5. Queste due proteasi giocano un ruolo essenziale poiché tagliano le due grandi poliproteine nelle proteine funzionali più piccole.
La protease principale di CoV-2, che effettua il maggior numero di tagli, pesa 33,8 kDa e si chiama Mpro, altrimenti conosciuta come proteasi 3C-simile (simile alla chimotripsina). La Mpro è fondamentale per la replicazione virale ed è assente nelle cellule umane. Per questa ragione essa rappresenta un buon bersaglio per lo sviluppo di nuovi farmaci antivirali: il blocco delle sue funzioni sarebbe infatti letale per il virus, ma sicuro per gli esseri umani.
Il meccanismo di azione di Mpro è simile a quello di altre proteasi. Tutte le proteasi possiedono due residui amminoacidici chiave: un residuo attivatorio (di solito un’istidina, His) che rimuove protoni da un gruppo ossidrilico o tiolico della catena laterale di un secondo residuo (di solito una serina, Ser, o una cisteina, Cys) che agisce come potente nucleofilo, ossia un potente donatore di elettroni. Nell’Mpro, che è una proteasi in cisteina, la diade catalitica è costituita da His41 e Cys1456 (i numeri identificano la posizione all’interno della sequenza amminoacidica della proteina).
Il meccanismo di reazione con cui Mpro catalizza l’idrolisi della poliproteina virale è descritto nella Figura 2.
Figura 2 Meccanismo della catalisi della cisteina proteasi Mpro.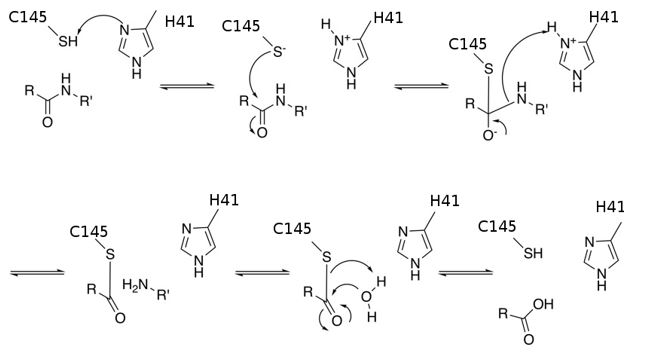
La catalisi inizia con la deprotonazione del tiolo (sostanzialmente un alcol in cui l’atomo di ossigeno è sostituito da un atomo di zolfo) della Cys145 da parte dell’His41, che è seguita dall’attacco nucleofilico della cisteina de-protonata al carbonio del legame peptidico della proteina substrato. Viene quindi rilasciata una proteina virale più piccola con terminale amminico libero, il residuo di istidina, His41, della proteasi recupera la sua forma de-protonata e si forma un intermedio tioestere che collega il nuovo carbossi-terminale del substrato al tiolo Cys145. Successivamente, si forma un tioestere intermedio che lega il nuovo terminale carbossilico al tiolo della Cys145. Il tioestere è quindi idrolizzato per generare un terminale carbossilico sulla poliproteina rimanente, rigenerando l’enzima libero. I residui Cys145 (C) e His41 (H) coinvolti nella reazione catalitica vengono indicati con codice a una lettera.
La struttura cristallografica della proteina Mpro in complesso con l’inibitore N3
Jin e collaboratori hanno ottenuto la struttura cristallografica ad alta risoluzione della proteina Mpro in complesso con N3 (Figura 3), una molecola nota per legare e inibire la proteasi principale di altri coronavirus, come SARS-CoV e MERS-CoV (codice di accesso in PDB: 6lu7)6. L’N3 è un inibitore simile a un oligopeptide (Figura 3D).
L’analisi della struttura mostra che la proteina appare come dimero (Figura 3A), formato da due subunità identiche di 306 amminoacidi (A e B). Ogni monomero è formato da tre differenti domini (Figura 3B):
- dominio I (residui 1–101);
- dominio II (residui 102–184);
- dominio III (residui 201–306) .
I domini I e II hanno una tipica struttura a barile β in cui i filamenti β si dispongono in modo antiparallelo, mentreil dominio III è formato da cinque α-eliche. Il dominio III è unito al dominio II da un lungo ripiegamento (residui 185–200). Il sito di legame al substrato consiste in una cavità profonda che è posta in prossimità dell’interfaccia del dimero tra i domini I e II e contiene la diade catalitica Cys145-His41 (Figura 3C). L’analisi della struttura ha consentito di capire che l’inibitore N3 lega fortemente la cavità dell’Mpro che normalmente alloggia il substrato, formando un legame covalente con la Cys145. L’analisi dell’interazione tra N3 e i residui che rivestono la cavità hanno fornito importanti informazioni strutturali per progettare inibitori potenti e reversibili dell’Mpro.
Come la Mpro di SARS-CoV, quella di CoV-2 taglia le poliproteine pp1a e pp1ab in specifiche posizioni amminoacidiche, identificando i siti di taglio grazie a particolari “sequenze di base” nelle poliproteine7. Le posizioni dei residui che appartengono alle sequenze di base nelle poliproteine sono nominate a seconda della posizione relativa rispetto al sito di taglio ed è possibile identificarle in modo molto specifico (per i dettagli, vedere la legenda della Figura 3C e la referenza bibliografica n. 8).
La sovrapposizione di sequenze delle Mpro di 12 coronavirus, inclusi CoV-2, SARS-CoV e MERS-CoV, mostra che i residui che rivestono la tasca che lega il substrato sono fortemente conservati9-12. Questo suggerisce che un potente inibitore di CoV-2 potrebbe essere un composto leader per guidare la sintesi di farmaci ad ampio spettro contro tutte le infezioni da coronavirus.
Figure 3 Struttura cristallografica della proteina Mpro del CoV-2 in complesso con l’inibitore N3.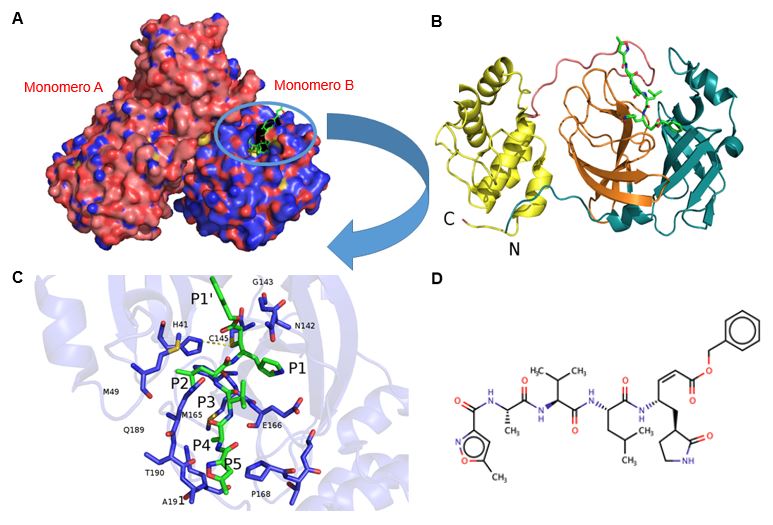
A Rappresentazione di superficie dell’omodimero di Mpro. Gli atomi di carbonio dei due monomeri A e B sono colorati rispettivamente in rosa salmone e blu chiaro. Gli atomi di azoto sono colorati in blu, quelli di ossigeno in rosso e quelli di zolfo in giallo. L’inibitore N3 è rappresentato con la struttura a bastoncini ed è colorato in verde. B Rappresentazione “a nastro” del monomero Mpro . I doimini I, II e III sono colorati, rispettivamente, in verde chiaro, arancio e giallo. La molecola N3 è colorata in verde. C Ingrandimento del sito di legame per N3. I residui catalici (H41 e C145) e quelli che interagiscono con l’N3 sono rappresentati come bastoncini blu. La posizione P1 corrisponde al residuo immediatamente prima del sito di taglio, seguito da P2, P3, P4, P5, fino alla parte N-terminale del sito di taglio. I siti corrispondenti in Mpro che interagiscono con i residui P1–P5 e P1′ sono denominati S1, S2, S3, S4, S5 and S1′. D Formula strutturale dell’inibitore N3 (N-[(5-Metilisossazol-3-yl)carbonil]alanil-L-valil-N~1~-((1R,2Z)-4-(Benzilossi)-4-osso-1-{[(3R)-2-ossopirrolidin-3-il]metil}But-2-enil)-L-Leucidammide). [Immagini adattate dalle strutture proteiche depositate nella PDB]
Dalla struttura dell’Mpro del CoV-2 ai farmaci: il ruolo degli screening virtuali e high-throughput
La struttura dell’Mpro di CoV-2 in complesso con l’inibitore N3 fornisce un modello e delle informazioni che possono essere usati per identificare altre molecole organiche capaci di legare la tasca catalitica dell’Mpro con più alta affinità, e condurre così allo sviluppo di nuovi farmaci antivirali specifici per il CoV-2. Una delle metodiche che è usata correntemente per identificare nuovi composti capostipiti è lo screening virtuale. Lo screening virtuale è una tecnica computazionale che permette di analizzare grandi numeri di dati (highthrouput screening) di librerie di migliaia di composti chimici, per identificare molecole che abbiano maggiori probabilità di legare il bersaglio farmacologico. Il legame del composto chimico al bersaglio viene simulato in silico (ossia in maniera totalmente predittiva), riducendo fortemente i costi, il tempo e lo sforzo rispetto allo stesso screening eseguito sperimentalmente. Una volta identificato un numero più piccolo di composti, vengono eseguiti esperimenti di laboratorio per confermare e ulteriormente selezionare il composto che lega a maggior affinità il bersaglio.
Jin e collaboratori6 hanno usato proprio questa tecnica per identificare molecole con un volume corrispondente a quello della tasca catalitica dell’Mpro, partendo dalla sua struttura cristallografica in complesso con N3. Tutti i composti, tipicamente piccole molecole organiche, conservate in una specifica banca dati, sono stati testati in silico per individuare quelli che, date le proprietà geometriche e chimiche, potessero essere potenzialmente in grado di legare la Mpro nella tasca catalitica. Lo screening dei composti viene effettuato tramite programmi dedicati. In questo caso è stato usato Glide (versione 8.2). Questa analisi ha portato all’identificazione di un composto, detto cinaserina, che ha mostrato il punteggio più alto e la modalità più sensata di legame alla tasca catalitica dell’Mpro. La cinaserina è un antagonista della serotonina molto ben caratterizzato, che è stato testato preliminarmente sugli esseri umani negli anni ’60 del Novecento e che ha mostrato di inibire l’Mpro di SARS-CoV. Di rilievo è il fatto che la cinaserina non è tossica per le cellule umane, mostrando una citotossicità del 50% a concentrazioni più alte di 200 μM, mentre è capace di inibire l’Mpro di CoV-2 a concentrazioni più basse (IC50=125 μM). Perciò, la cinaserina rappresenta un buon candidato come farmaco antivirale, che potrebbe essere ottimizzato per ottenere il farmaco nella sua forma finale13.
Infatti, una volta che un nuovo composto è identificato, viene inizialmente somministrato ad animali da esperimento, e successivamente agli esseri umani nel corso di sperimentazioni cliniche per testarne sicurezza ed efficacia. Possono volerci dai 10 ai 15 anni, o anche di più, per completare tutte le fasi di una sperimentazione clinica prima di giungere allo stadio di licenza di un nuovo farmaco. Una strategia alternativa per accelerare i tempi è quella di testare l’attività di molecole esistenti e già approvate per altre patologie. Jin e collaboratori hanno applicato questa strategia per trovare velocemente farmaci contro COVID-1914. Infatti, hanno analizzato oltre 10 000 composti come ligandi possibili della Mpro, inclusi farmaci già approvati, alcuni candidati per le sperimentazioni cliniche e anche dei prodotti naturali. Tra questi, hanno identificato un derivato del 5-fluorouracile, il Carmofur (1-hexylcarbamoyl-5-fluorouracil), che è risultato capace di legare ed inibire l’Mpro (Figura 4, a sinistra). Il Carmofour è stato già approvato come farmaco antitumorale ed è usato per trattare il cancro del colon retto fin dal 1980. Sempre, Jin e collaboratori hanno risolto la struttura cristallografica dell’Mpro in complesso con il Carmofour, scoprendo le basi molecolari dell’efficacia del composto. In particolare, l’acido grasso di Carmofour lega la Cys145 attraverso un legame covalente, inibendo l’enzima (Figura 4, a destra). Questo studio è particolarmente importante poiché dimostra che il Carmofour, un farmaco già approvato e commercialmente disponibile, potrebbe essere potenzialmente usato contro la COVID-19 e fornisce le basi strutturali per progettare nuovi e più potenti inibitori ad ampio spettro che potrebbero essere usati per trattare tutte le infezioni da coronavirus.
Figura 4 Ingrandimento del sito catalitico di Mpro legato a Carmofour.
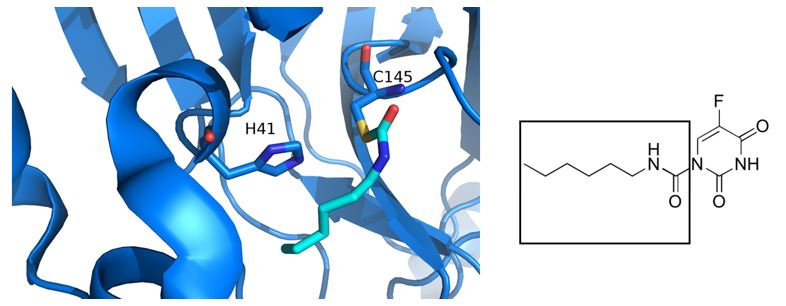
A sinistra, Carmofour all’interno dll’Mpro. A destra è rappresentata la struttura chimica di Carmofour; nel riquadro è indicato l’acido grasso (C7H14NO) che lo compone e che forma un legame covalente di tipo tioestere con la C145 dell’Mpro. [Immagine a sinistra: PDB: 7BUY]
La proteina Spike del CoV-2: la chiave molecolare del virus
I virus evolvono continuamente le proteine della loro superficie per potenziare l’interazione con i recettori sulle cellule ed entrare in esse con maggior efficienza secondo il modello chiave-serratura. Questo è anche il caso della proteina Spike (S) di CoV-2 (la chiave) e del recettore umano Angiotensin Converting Enzyme 2 (hACE2, la serratura).
La proteina S è una delle più interessanti e studiate tra quelle che contribuiscono al legame con il recettore dell’ospite e alla patogenesi virale. La proteina S “decora” la superficie del virus ed è responsabile per l’aspetto a corona della superficie virale, da cui il nome coronavirus. Questa è usata dal virus come una chiave per entrare nelle cellule ospite15. Agisce legando il recettore sulle cellule bersaglio, induce l’endocitosi dei virioni e catalizza la fusione tra le membrane cellulari e virali, assicurando l’ingresso dell’RNA genomico virale nel citoplasma delle cellule. La proteina S rappresenta anche il bersaglio principale del sistema immunitario, attivandolo e inducendo la produzione di anticorpi. Per questa ragione è considerata il bersaglio primario di farmaci antivirali e vaccini. Fortunatamente, la mole di dati strutturali sulle proteine S dei coronavirus correlati al SARS-CoV (SARSr-CoVs, SARS-related CoVs), inclusa la struttura recentemente determinata di S di CoV-2 (d’ora in avanti indicata con CoV-2-S), costituiscono una ricca fonte di informazioni utili per progettare molecole inibitrici della sua funzione e, quindi, potenzialmente usabili intrattamenti terapeutici.
L’organizzazione strutturale di CoV-2-S è molto simile a quella delle proteina S di altri coronavirus come SARS-CoV e MERS-CoV. Essa è una proteina trimerica transmembrana formata da tre unità identiche, dette protomeri (Figura 5). Ogni protomero espone all’esterno delle catene di zuccheri che servono a ingannare il sistema immunitario16,17.
Figura 5 Rappresentazione della struttura della CoV-2-S.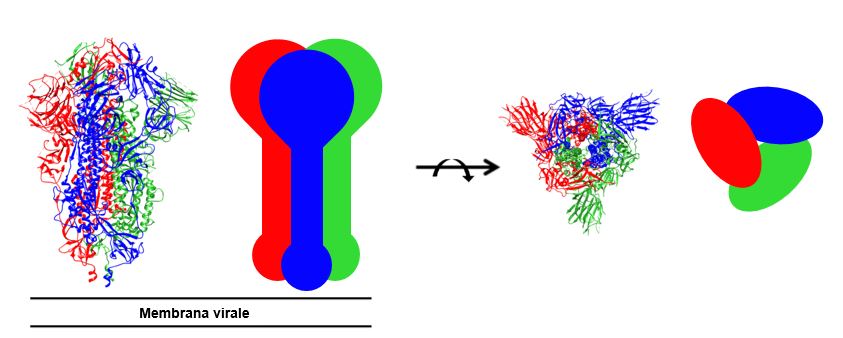
La struttura della CoV-2-S è formata da tre proteine identiche (protomeri, rappresentati rispettivamente in verde, rosso e blu) assemblate in un trimero. [Immagini adattate da: PDB: 6VXX]
Ogni protomero di CoV-2-S (per esempio, quello blu nella Figura 5) comprende due subunità funzionali: una responsabile per il legame al recettore sulle cellule bersaglio (la subunità S1) e l’altra coinvolta nella fusione con la membrane cellulare (subunità S2). Più in dettaglio, la subunità S1 contiene:
- un dominio N-terminale (NTD, N-Terminal Domain);
- due subdomini detti SD1 e SD2 (Subdomain 1 e 2);
- il dominio di legame al recettore (RBD, Receptor Binding Domain) responsabile per il legame alla cellula ospite attraverso l’interazione con ACE2 (vedi oltre);
- un dominio C-terminale che contiene il macchinario di fusione che aiuta il virus ad entrare nelle cellule (Figura 6A)16,17.
La CoV-2-S, come varie proteine spike di altri SARSr-CoV, è tagliata da proteasi cellulari al confine tra le subunità S1 ed S2, generando due regioni separate che rimangono legate in modo non covalente nella cosiddetta “conformazione di prefusione”. Infatti, CoV-2-S esiste in due differenti conformazioni, chiamate “su” (up) e “giù” (down) (Figura 6B). Nella conformazione “giù”, la CoV-2-S non può mediare la fusione della CoV-2 con la membrana della cellula ospite. Per assicurare l’ingresso del virus, deve andare incontro ad un cambiamento conformazionale, che garantisce l’acquisizione della conformazione “su”16.
Figura 6 Struttura primaria della CoV-2-S e delle conformazioni giù/su del dominio RBD.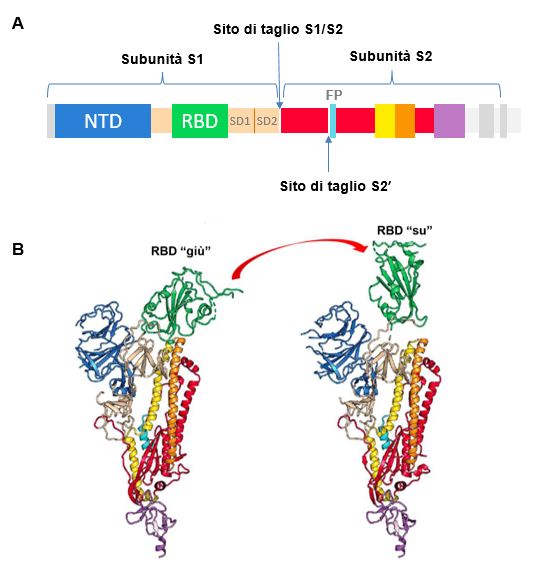
A Schema della struttura primaria della CoV-2-S con indicati i domini e i siti di taglio. B Un singolo protomero di CoV-2-S che mostra l’RBD nella conformazione “giù” o “su” (up, right). [Basato su Wrapp et al. 16]
L’acquisizione della conformazione “su” si verifica non appena la CoV-2-S si avvicina ad ACE2; in seguito, avviene il taglio da parte delle proteasi cellulari nel sito S1/S2 (Figura 7). Dopo il taglio, le subunità S1 e S2 assolvono a differenti funzioni:
- la S1 lega ACE2 attraverso l’RBD;
- la S2 media l’ingresso del virus dopo un ulteriore taglio a un sito secondario chiamato S2′. Questo taglio si pensa attivi la fusione di membrane attraverso un altro esteso ed irreversibile cambiamento conformazionale.
In conclusione, l’ingresso del coronavirus in cellule suscettibili è un processo complesso che richiede l’azione concertata del legame al recettore e del processamento proteolitico della proteina S per promuovere la fusione tra virus e cellula. Una delle proteasi cellulari coinvolta nell’ingresso della CoV-2 nelle cellule ospiti è la serina proteasi transmembrana 2 (TMPRSS2, Transmembrane Serine Protease 2), che è richiesta anche per l’infezione da SARS-CoV. Infatti, l’inibizione di TMPRS22 blocca l’ingresso di SARS-CoV e CoV-2 nelle cellule.
È interessante il fatto che la CoV-2-S possieda un’inserzione di quattro amminoacidi al confine tra S1 ed S2 rispetto a alla proteina S di SARS-CoV. Questi quattro amminoacidi addizionali costituiscono il sito di taglio per una specifica proteasi umana chiamata furina16. La presenza di questo sito di taglio peculiare per la furina in CoV-2-S ha fatto ipotizzare che, data l’espressione praticamente ubiquitaria delle proteasi simili alla furina, esse potrebbero aver partecipato all’acquisizione del più ampio tropismo cellulare e tissutale di CoV-2 rispetto a SARS-CoV, come anche a un incremento della sua trasmissibilità e patogenicità16. È stato dimostrato di recente che, come TMPRSS2, la furina sia essenziale per l’ingresso di CoV-2 nelle cellule ospiti. Inoltre anche la catepsina D, una proteasi tipica dei lisosomi, è richiesta per un ingresso efficiente del CoV-2. Inoltre, è di rilievo che tutte queste proteasi sembrino cooperare per mediare l’infezione da CoV-2. Questo non è il caso di SARS-CoV, che non ha un sito di taglio per la furina.
Figura 7 Rappresentazione schematica della CoV-2-S e i siti di taglio per la furina e la TMPRSS2.
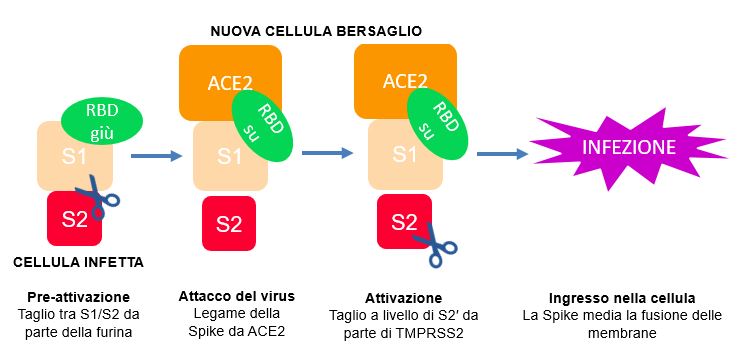
Nelle cellule infette o durante la biosintesi, la furina taglia la CoV-2-S nel sito S1/S2. Poi la CoV-2-S media l’attacco del virus alle cellule ospite. Per entrare in maniera efficace nelle cellule, la CoV-2-S ha bisogno di essere attivata dalla TMPRSS2.
Aspetti strutturali dell’interazione tra CoV-2-S e ACE2: la porta d’ingresso per l’invasione delle cellule umane
Coronavirus differenti usano diversi domini di S1 per riconoscere i vari “siti di attracco” e recettori di ingresso. Le proteine Spike di CoV-2 SARS-CoV S condividono lo stesso recettore, ACE217, 18, 19. Il recettore ACE2 è coinvolto nel mantenimento dell’omeostasi della pressione sanguigna, nel bilancio degli elettroliti e nella risposta infiammatoria ed è richiesto per il corretto funzionamento di rene, cuore, vasi sanguigni e polmoni; infatti esso idrolizza e converte l’angiotensina II in molecole vasodilatatorie e cardioprotettive. Le proteine ACE2 e CoV-2-S co-localizzano sulla membrana delle cellule ospite dove interagiscono l’una con l’altra. Recentemente, è stata determinata la struttura cristallografica del complesso tra l’RBD di CoV-2-S ed ACE2 (Figura 8) 20, 21.
Figura 8 La CoV-2-S in complesso con l’ACE2 umano.
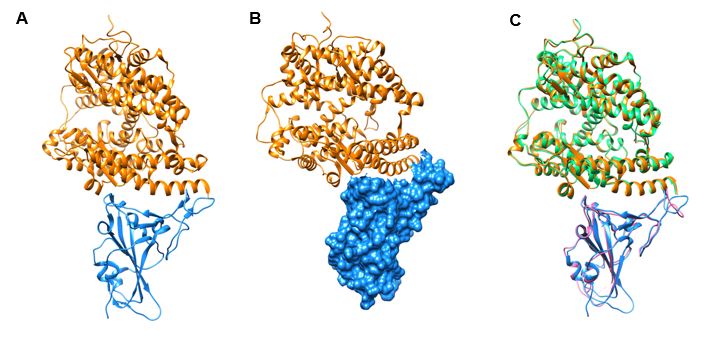
A Struttura complessiva dell’RBD della CoV-2-S (azzurro) legato all’ACE2 (arancio). B Diverso orientamento del complesso con l’RBD (rappresentato con il modello di superficie) della CoV-2-S. C Sovrapposizione della struttura dell’RBD della CoV-2-S legato ad ACE2 e della struttura dell’RBD del CoV-S (in verde) legato all’ACE2 (in rosa). [Immagine da PDB: 6M0J,21]
Come si può osservare dalla Figura 8, il recettore ACE2 interagisce con il dominio RBD della CoV-2-S essenzialmente grazie a un’α-elica che si accomoda dentro una tasca della CoV-2-S. Una piccola porzione dell’RBD di Spike interagisce con ACE220, 21. Studi strutturali hanno consentito di mappare nella struttura della CoV-2-S i residui che interagiscono con l’ACE2 e di compararli con l’RBD della CoV-S, rivelando una modalità di legame simile. I dettagli a livello atomico dell’interfaccia del complesso hanno mostrato che l’interazione tra CoV-2-S ed ACE2 è mediata da un più alto numero di amminoacidi rispetto all’interazione tra CoV-S e ACE2: 21 contatti sono stati trovati nel complesso CoV-2-S/ACE2 e 17 nel complesso CoV-S/ACE2. Sostituzioni di residui chiave in CoV-2-S rafforzano leggermente l’interazione, portando, probabilmente, a una più alta affinità per il recettore rispetto al SARS-CoV-S21.
Porte di accesso alternative: le integrine e il CD147
Un’altra caratteristica interessante della CoV-2-S è la presenza, sempre nell’RBD ma a monte del dominio di interazione con ACE2, di una sequenza di amminoacidi specifici, detto motivo RGD (R = arginina; G = glicina; D = acido aspartico; Figura 9) riconosciuto da un’altra classe di recettori, le integrine22. Le integrine sono eterodimeri ubiquitari, espressi sulla superficie cellulare, che mediano l’adesione cellulare, la migrazione e attivano varie vie di segnalazione intracellulari. Nella conformazione “su”, il motivo RGD di CoV-2-S è esposto sulla superficie, mentre nella conformazione “giù” è nascosto (Figura 8). Questo motivo RGD è unico nel CoV-2 tra i β-coronavirus, mentre è presente negli adenovirus e nello metapneumovirus umani, che usano le integrine come vie di ingresso nelle cellule. Questa sequenza potrebbe essersi evoluta come meccanismo alternativo di ingresso del virus o per ampliare il suo tropismo. Inoltre, è stato dimostrato che anche il CD147, usato dal SARS-CoV come recettore di ingresso alternativo, potrebbe essere usato anche dal CoV-2, poiché un anticorpo diretto contro il CD147 impedisce l’ingresso del CoV-2 nelle cellule23.
Figura 9 Motivo RGD.
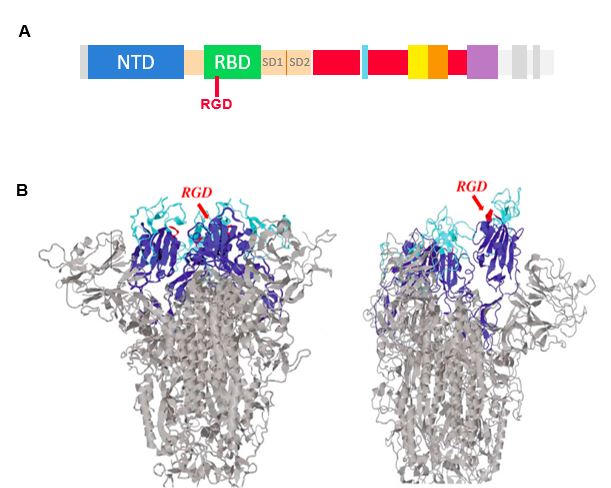
A La sequenza RGD è a monte del dominio di legame all’ACE2. B Struttura della CoV-2-S nella conformazione “giù” o “su”: la sequenza RGD (in rosso) è nascosta quando la Spike non può legare l’ACE2 ed esposta quando la Spike lega l’ACE2 [Adattato da Sigrist et al., Elsevier 2020 Licenza Creative Commons CC-BY]
L’RNA polimerasi RNA dipendente (RdRp) del CoV-2: la macchina di replicazione del virus
Nel maggio 2020, vari laboratori in Cina, in particolare di Shangai, e Germania hanno pubblicato la struttura di una proteina del CoV-2 che potrebbe rappresentare un terzo bersaglio importante per il trattamento dell’infezione. Essi hanno studiato la struttura al criomicroscopio elettronico (cryo-EM) della RdRp, una componente chiave del macchinario di replicazione del virus24, 25, 26.
La replicazione dell’RNA positivo di CoV-2 (v. Il mondo sorprendente del genoma di SARS-CoV-2) è mediata da un complesso multiproteico, composto da proteine non strutturali (nsp, non-structural protein), codificate dal virus ma espresse solo nelle cellule infette. Queste includono vari enzimi e fattori di trascrizione che assicurano al virus le capacità di replicarsi e produrre molte copie di se stesso27. La componente di base di questo complesso è la subunità catalitica nsp12. Da sola, nsp12 ha poca attività e capacità di legare l’RNA stampo: per attivarsi ha bisogno di legare i fattori accessori nsp7 ed nsp828. Dato il ruolo chiave di questo macchinario molecolare nella replicazione virale, la polimerasi RdRp è stata proposta come bersaglio di una classe di farmaci antivirali, costituiti da analoghi di nucleosidi (v. anche La lunga strada verso le terapie). Tra questi, il remdesivir merita di essere menzionato. Il remdesivir è un cosiddetto profarmaco che deve essere convertito nella sua forma attiva all’interno delle cellule, dove assume la forma trifosfato (RTP, Remdesivir TriPhosphate)29. Perciò, l’RdRp è stata oggetto di un intenso studio strutturale che ha consentito la determinazione della struttura al criomicroscopio elettronico (cryo-EM) del complesso nsp12-nsp7-nsp8 del CoV-2, fornendo la struttura complessiva della RdRp (Figura 10). La RdRp di CoV-2 è stata anche studiata in complesso con un RNA stampo e con il remdesivir, rivelando le basi molecolari del meccanismo di inibizione del farmaco, come spiegato nel dettaglio più avanti nel testo.
Figura 10 Struttura della RdRp.
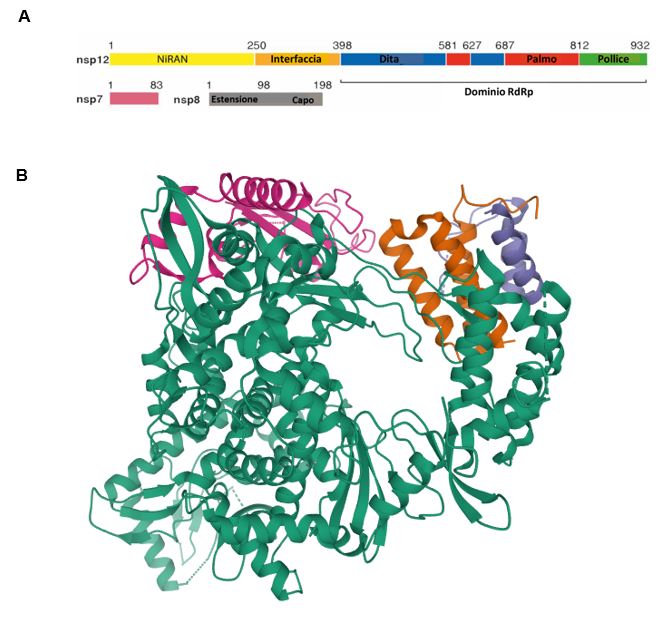
A Struttura dei domini delle subunità di RdRp nsp12, nsp8, and nsp725. B Struttura tridimensionale della RdRp ottenuta con la criomicroscopia elettronica. Si veda anche la ricostruzione in cryo-EM nella Figura 1 dell’articolo di Gao et al., 2020 e la Figura 11. [Immagine in B da PDB: 6M71]
Nel 2019 è stata determinata la struttura cryo-EM di RdRp del SARS-CoV28. La RdRp del SARS-CoV e del CoV-2 hanno sequenze amminoacidiche simili, mostrando complessivamente la stessa struttura (Figura 11) e un identico sito attivo che catalizza la polimerizzazione dell’RNA (Figura 12). La nsp12 contiene il dominio NiRAN (Nidovirus RdRp-Associated Nucleotydil-transferase domain, dominio nucleotidil-trasferasico associato alle RdRp di nidovirus) tipico di tutti i coronavirus30. Il dominio nps12 mostra la conformazione canonica a mano a coppa destrorsa, con subunità che possono essere rappresentate come dita, palmo e pollici, con le dita che formano un cerchio chiuso con i pollici (Figura 11). La nsp12 lega il primo giro dell’RNA tra le dita e il pollice e il sito attivo è posto nel palmo.
Le interazioni tra proteina ed RNA coinvolgono lo scheletro dell’RNA (formato da ribosio-fosfato), assicurando la distinzione tra DNA (che contiene deossiribosio-fosfato) e RNA. Il legame dell’RNA sembra essere indipendente dalla sequenza, poiché non ci sono contatti con le coppie di basi dell’RNA. L’RNA a doppia elica uscente contatta le lunghe α-eliche formate dalle regioni N-teminali delle due subunità nsp8. Queste lunghe α-eliche sono mobili quando l’RdRp è libera, indicando che l’nsp8 ha estensioni flessibili che diventano ordinate solo quando un RNA a doppio filamento fuoriesce dall’enzima. Esse agiscono come uno scivolo che guida l’RNA uscente ed evitano la sua dissociazione dall’RdRp durante la replicazione (Figura 11). La molecola di remdesivir monofosfato (RMP,Remdesivir MonoPhosphate) si localizza al centro del sito attivo ed è legata in maniera covalente al filmaneto di innesco in posizione +1 (a livello della prima base). Questa osservazione ha fatto nascere l’idea che questo farmaco agisca come terminatore della sintesi del filamento di RNA, come altri analoghi dei nucleosidi (Figura 12, v. anche La lunga strada verso le terapie).
Figura 11 RdRp complessata con l’RNA a doppio filamento.
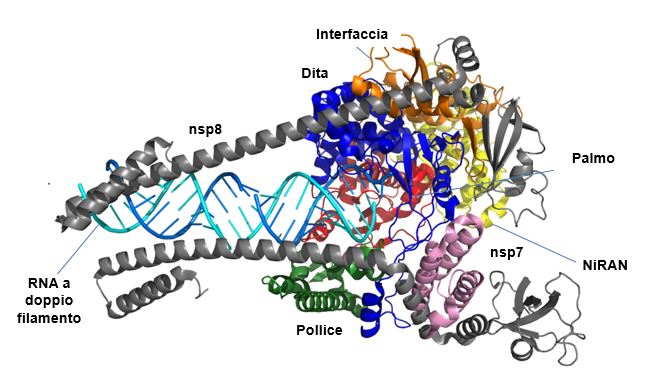
Visionedall’alto della struttura delle subunità nsp12, nsp8 ed nsp7 di RdRp, in cui è possibile notare l’RNA a doppio filamento (in azzurro chiaro e scuro) che sembra scivolare sulle lunghe eliche della proteina nsp8 (codice colore: nsp8 in grigio, pollice in verde, palmo in rosso, dita in blu, NiRAN in giallo, interfaccia in arancio). [Immagine da PDB: 6YYT]
Figura 12 Ingrandimento del sito attivo di RdRp.
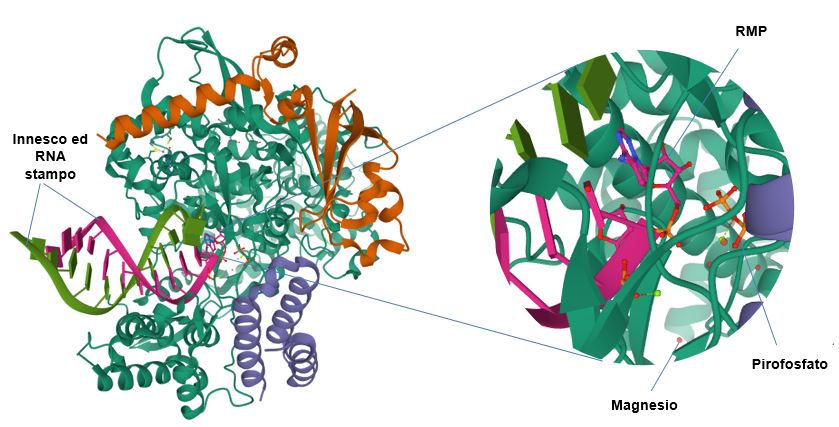 Ingrandimento del sito attivo di RdRp, che mostra il remdesivir monofosfato (RMP, Remdesivir MonoPhosphate), il pirofosfato e gli ioni magnesio. [Immagine: PDB: 7BV2]
Ingrandimento del sito attivo di RdRp, che mostra il remdesivir monofosfato (RMP, Remdesivir MonoPhosphate), il pirofosfato e gli ioni magnesio. [Immagine: PDB: 7BV2]
APPROFONDIMENTI: LE TECNICHE
Criomicroscopio a trasmissione elettronica: la macchina potente che fotografa le grandi biomolecole
La microscopia elettronica è simile alla microscopia ottica: entrambe producono immagini ingrandite di piccole aree di materiale biologico per distinguerne i dettagli, ma un microscopio elettronico usa un fascio di elettroni accelerati anziché fotoni come fonte di radiazione. Analogamente alle lenti di vetro di un microscopio ottico, la colonna ottica elettronica è costituita da una serie di lenti magnetiche, composteda campi elettrici e non da materiale solido. Queste lenti mettono a fuoco e controllano la posizione del fascio di elettroni emessi da un cannone elettronico per ottenere l’immagine delle molecole. .
Nella microscopia elettronica a trasmissione (TEM, Transmission Electron Microscopy), gli elettroni sono trasmessi attraverso l’oggetto.
Gli elettroni trasmessi impressionano una pellicola o una camera digitale, rappresentando la densità elettronica dell’oggetto di interesse. Il vantaggio più prezioso dell’uso degli elettroni come fonte di radiazione è la più alta risoluzione che teoricamente è possibile ottenere. Il termine risoluzione identifica la distanza minima alla quale due oggetti possono essere distinti. Con un microscopio ottico è possibile raggiungere una risoluzione di circa 0,2 μm, che consente di osservare singole cellule, ma non oggetti più piccolo come particelle virali, proteine e altre molecole biologiche. Poiché la lunghezza d’onda di un fascio di elettroni accelerati è circa 100 000 volte più piccolo della luce visibile, che corrisponde a qualche unità di picometri (10−12 m), la risoluzione che in principio può essere raggiunta con un microscopio elettronico è teoricamente illimitata. In pratica, la risoluzione in microscopia elettronica è limitata a circa 1 Angstrom (1 Å = 10−10 m; è un’unità di distanza usata per misurare il raggio atomico e le distanze interatomiche) a causa delle aberrazioni nelle lenti ed altri problemi tecnici legati al microscopio, ma può essere usata per visualizzare particelle virali (≤ 100 nanometri) o proteine (100-10 nanometri) e per studiare molecole a risoluzione atomica.
La comune metodica di microscopia elettronica per ottenere immagini a contrasto di specie biologiche consiste nell’immergere per un tempo limitato il campione in coloranti costituiti da sali di metalli pesanti: poiché il colorante penetra solo nella griglia di supporto su cui è posizionato il materiale biologico e non nelle particelle del campione, quest’ultimo risulta un oggetto chiaro su sfondo scuro (colorazione negativa). Comunque, la risoluzione massima che può essere ottenuta mediante la tecnica della colorazione negativa è dell’ordine dei 40–20 Å. In anni più recenti, l’introduzione di condizioni “a freddo” (“crio”, −190 °C) nella preparazione dei campioni e di tecnologie avanzate per il rilevamento degli elettroni e per l’analisi dei dati, ha causato una rivoluzione nel campio della microscopia elettronica, che è diventata una tecnica all’avanguardia estremamente potente e capace di fornire informazioni strutturali a risoluzione atomica di molecole che hanno rappresentato a lungo il “Santo Graal” della biologia strutturale. Nella criomicroscopia elettronica (cryo-EM) il campione, assorbito e congelato istantaneamente su un supporto poroso conserva la sua struttura nativa e idratata all’interno del ghiaccio vitreo che si forma sui buchi della pellicola di supporto e che può essere visualizzato senza colorazione. L’oggetto (la biomolecola), orientata a caso nello spazio, è, quindi, fotografata. Vengono poi raccolte molte immagini in due dimensioni della particella vista a 360°. Queste immagini 2D vengono usate per ricostruire, tramite sofisticati algoritmi, la struttura tridimensionale della molecola. Oggi, il maggior limite di questa tecnica è la grandezza della molecola che può essere osservata, rendendo la metodica della cryo-EM una scelta d’elezione per lo studio di grandi complessi macromolecolari.
La cryo-EM, accoppiata con una tecnica di processamento delle immagini, chiamata analisi a singola particella, ha rivoluzionato il nostro modo di studiare i sistemi biologici e negli ultimi mesi ha consentito in pochissimo tempo la visualizzazione in pochissimo tempo della struttura e dell’architettura di componenti macromolecolari del nuovo coronavirus.
Cristallografia a raggi X
La struttura tridimensionale delle macromolecole biologiche ci consente di comprendere i meccanismi alla base dei processi biologici in cui sono coinvolte e di descrivere le interazioni atomiche che li guidano, ovvero di capire come una particolare macromolecola possa svolgere la propria funzione. La cristallografia a raggi X è un’altra metodologia sperimentale che permette di determinare la distribuzione nello spazio degli atomi di una molecola presente in un cristallo, analizzando come i raggi X vengono sono diffratti da quest’ultimo ed è molto utile per determinare la struttura tridimensionale a risoluzione atomica di macromolecole biologiche. Perché due oggetti possano essere visualizzati come entità distinte, la lunghezza d’onda della radiazione elettromagnetica usata deve essere della stessa grandezza della distanza che intercorre tra gli stessi oggetti. Poiché le distanze interatomiche sono tipicamente di circa i 1Å e la lunghezza d’onda dei raggi X cade nell’intervello compreso tra 0,1 e 2 Å, è possibile rivelare la struttura atomica delle molecole usando una radiazione elettromagnetica a raggi X.
Il problema più arduo di questa tecnica risiede nella mancanza di lenti ottiche capaci di catturare i raggi X. Perciò, sono stati sviluppati metodi indiretti e piuttosto complicati per ottenere le strutture delle macromolecole partendo dalle immagini di diffrazione della radiazion X.
Il termine cristallografia deriva dalla necessità per questa tecnicadi produrre un cristallo a partire da macromolecole in soluzione (il cristallo è un materiale solido composto da un distribuzione altamente ordinate di un gran numero di molecole ripetute periodicamente nelle tre dimensioni spaziali),. La diffrazione di una molecola singola originerebbe infatti un segnale non rilevabile. Un cristallo, invece, che è composto da un gran numero ampio di copie della stessa molecola ripetute nelle tre dimensioni in maniera ordinata e periodica, agisce come un amplificatore di segnale e in determinate condizioni, note come condizioni di interferenza costruttiva, le onde diffratte dalle molecole di cristallo si addizionano e rendono rilevabile il segnale totale di diffrazione.
Se esposti a una sorgente di raggi X, i cristalli originano delle figure (pattern) di diffrazione che contengono molte, ma non tutte, le informazioni richieste per determinare la struttura tridimensionale delle macromolecole che costituiscono il cristallo oggetto di analisi. Tuttavia, partendo dal pattern di diffrazione e usando metodiche diverse e strumenti computazionali per analizzarlo, è possibile ottenere la mappa di densità elettronica della macromolecola che è, quindi, utilizzata per costruire il suo modello atomico tridimensionale.
Degno di nota è il fatto che cristallizzando la macromolecola in presenza di uno specifico ligando (per esempio un candidato farmaco) è anche possibile ottenere informazioni sull’interazione, fornendo elementi chiave per il miglioramento dell’interazione tra macromolecola e ligando per ottenere candidati ancor più efficaci da proporre per lo sviluppo di nuovi farmaci.
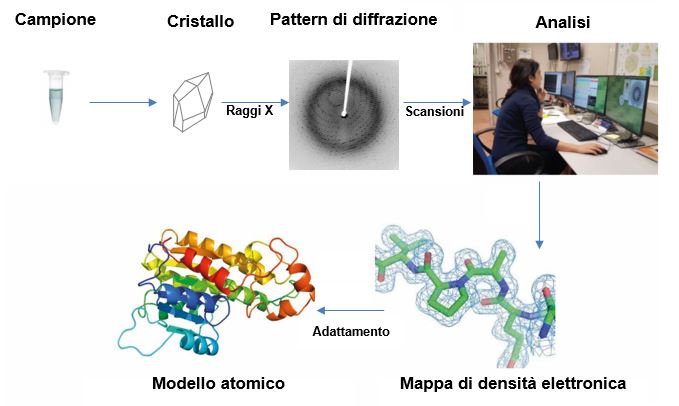
[Pattern di diffrazione: Dahl/Wikipedia CC-BY; strutture proteiche: PDB]
“Per i non esperti”
Protein Data Bank (PDB)
La Protein Data Bank (PDB), è una banca dati, accessibile gratuitamente a tutti, nella quale sono conservate le strutture tridimensionali di proteine e acidi nucleici. I dati, ottenuti generalmente tramite cristallografia a raggi X, spettroscopia a risonanza magentica nucleare o criomicroscopia elettronica, sono depositati dai laboratori di ricerca di tutto il mondo.
Proteasi
Le proteasi sono enzimi che tagliano altre proteine a livello dei legami peptidici, legami chimici tra il gruppo carbossilico di un amminoacido e il gruppo amminico di un altro.
Oligopeptide
Un oligopeptide è un peptide costituito da due a poche decine di amminoacidi.
Struttura a barile β
Una struttura a barile β è costituita da un foglietto β, formato da ripetizioni in tandem, che gira e si avvolge dando luogo a una struttura toroidale in cui il primo filamento è legato all’ultimo.
α-Elica
Un’α-elica è una struttura elicoidale, la più semplice struttura secondaria delle proteine.
IC
IC sta per concentrazione inibitoria; per una molecole, IC50 corrisponde alla metà della concentrazione massima con cui essa è capace di inibire il suo bersaglio ed è una misura della sua potenza inibitoria.
Modello dell’interazione chiave-serratura
Tutte le infezioni virali richiedono l’interazione tra particelle virali e la superficie della cellula ospite. Possiamo immaginare che le nostre cellule posseggano sulla loro superficie una serie di “serrature”, detti recettori, ognuna adatta, di solito, ad una “chiave” specifica, detta ligando, una proteina che può essere solubile o presente sulla superficie di cellule limitrofe. Quando la chiave entra nella serratura, altre proteine vengono attivate all’interno della cellula, lavorando in sequenza in una cascata di eventi che termina nel nucleo. Questa cascata di eventi culmina in un cambiamento nell’espressione di geni, che deve essere appropriato allo stimolo ricevuto. Per rendere le cose più chiare possibili, si può immaginare questo proceso come il meccanismo di accensione del motore di un’automobile, dove l’automobile è la cellula e il motore è il nucleo che governa i comportamenti della cellula: quando si gira la chiave nel nottolino dell’automobile, una serie di meccanismi permette al motore di avviarsi.
Un esempio è la risposta a proteine, come i fattori di crescita, che impartiscono alla cellula l’ordine di proliferare e dividersi in due cellule figlie.
Recettori
I recettori sono proteine di membrane esposte sulla superficie della cellula. Mediano:
- l’accesso alla cellula di altri fattori o proteine;
- la comunicazione tra l’esterno e l’interno della cellula, legando molecole e inviando segnali specifici, a cui segue l’attivazione di una risposta specifica.
I recettori sono anche sfruttati dai virus e usati come porte d’ingresso per iniziare l’infezione.
Bibliografia
- Colavita, F. et al. SARS-CoV-2 isolation from ocular secretions of a patient with COVID-19 in Italy with Prolonged viral RNA detection. Ann Intern Med, M20-1176, doi:10.7326/M20-1176 (2020).
- McBride, R., van Zyl, M. & Fielding, B. C. The Coronavirus nucleocapsid is a multifunctional protein. Viruses 6(8), 2991-3018, doi:10.3390/v6082991 (2014).
- General Office of National Health Commission; General Office of National Administration of Traditional Chinese Medicine. Diagnostic and treatment protocol for Novel Coronavirus Pneumonia (Trial version 6) (2020).
- Zhou, P. et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature 579(7798), 270-273 (2020).
- Wu, F. et al. A new coronavirus associated with human respiratory disease in China. Nature 579(7798), 265-269 (2020).
- Jin, Z. et al. Structure of Mpro from SARS-CoV-2 and discovery of its inhibitors. Nature 582(7811), 289-293 (2020).
- Kiemer, L., Lund, O., Brunak, S. & Blom, N. Coronavirus 3CLpro proteinase cleavage sites: possible relevance to SARS virus pathology. BMC Bioinformatics 5, 72, doi:10.1186/1471-2105-5-72 (2004).
- Berger, A. & Schechter, I. Mapping the active site of papain with the aid of peptide substrates and inhibitors Philos Trans R Soc Lond B Biol Sci 257(813), 249-264, doi:10.1098/rstb.1970.0024 (1970).
- Anand, K. et al. Structure of coronavirus main proteinase reveals combination of a chymotrypsin fold with an extra α-helical domain. EMBO J 21(13), 3213-3224, doi:10.1093/emboj/cdf327 (2002).
- Yang, H. et al. The crystal structures of severe acute respiratory syndrome virus main protease and its complex with an inhibitor. Proc Natl Acad Sci U S A 100(23), 13190-13195, doi:10.1073/pnas.1835675100 (2003).
- St John, S. E., Tomar, S., Stauffer, S. R. & Mesecar, A. D. Targeting zoonotic viruses: structure-based inhibition of the 3C-like protease from bat coronavirus HKU4—the likely reservoir host to the human coronavirus that causes Middle East respiratory syndrome (MERS). . Bioorg Med Chem 23(17), 6036-6048, doi:10.1016/j.bmc.2015.06.039 (2015).
- Wang, F. et al. Crystal structure of feline infectious peritonitis virus main protease in complex with synergetic dual inhibitors. J Virol 90(4), 1910-1917, doi:10.1128/JVI.02685-15 (2015).
- Chen, L. et al. Cinanserin is an inhibitor of the 3C-like proteinase of severe acuterespiratory syndrome coronavirus and strongly reduces virus replication in vitro. J Virol 79(11), 7095-7103, doi:10.1128/JVI.79.11.7095-7103.2005 (2005).
- Jin, Z. et al. Structural basis for the inhibition of SARS-CoV-2 main protease by antineoplastic drug carmofur Nat Struct Mol Biol 27(6), 529-532, doi:10.1038/s41594-020-0440-6 (2020).
- Tortorici, M. A. et al. Structural basis for human coronavirus attachment to sialic acid receptors. Nat Struct Mol Biol 26, 481-489, doi:10.1038/s41594-019-0233-y (2019).
- Wrapp, D. et al. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation. Science 367(6483), 1260-1263, doi:10.1126/science.abb2507 (2020).
- Walls, A. C. et al. Structure, function, and antigenicity of the SARS-CoV-2 Spike glycoprotein. Cell 181(2), 281-296.e286, doi:10.1016/j.cell.2020.02.058 (2020).
- Hoffmann, M. et al. SARS-CoV-2 cell entry depends on ACE2 and TMPRSS2 and is blocked by a clinically proven protease Inhibitor. Cell 181(2), 271-280.e278, doi:10.1016/j.cell.2020.02.052 (2020).
- Letko, M., Marzi, A. & Munster, V. Functional assessment of cell entry and receptor usage for SARS-CoV-2 and other lineage B betacoronaviruses. Nat Microbiol 5, 562–569, doi:10.1038/s41564-020-0688-y (2020).
- Lan, J. et al. Structure of the SARS-CoV-2 spike receptor-binding domain bound to the ACE2 receptor. Nature 581, 215-220, doi:10.1038/s41586-020-2180-5 (2020).
- Wang, Q. et al. Structural and functional basis of SARS-CoV-2 entry by using human ACE2. Cell, pii: S0092-8674(0020)30338-X, doi:10.1016/j.cell.2020.03.045 (2020).
- Sigrist, C. J., Bridge, A. & Le Mercier, P. A potential role for integrins in host cell entry by SARS-CoV-2. Antiviral Res 177, 104759, doi:10.1016/j.antiviral.2020.104759 (2020).
- Ulrich, H. & Pillat, M. M. CD147 as a Target for COVID-19 Treatment: Suggested Effects of Azithromycin and Stem Cell Engagement. Stem Cell Rev Rep 16, 434-440, doi:10.1007/s12015-020-09976-7 (2020).
- Yin, W. et al. Structural basis for inhibition of the RNA-dependent RNA Polymerase from SARS-CoV-2 by Remdesivir. Science 368(6498), 1499-1504, doi:10.1126/science.abc1560 (2020).
- Gao, Y. et al. Structure of the RNA-dependent RNA Polymerase from COVID-19 virus. Science 368(6492), 779-782, doi:10.1126/science.abb7498 (2020).
- Hillen, H. S. et al. Structure of replicating SARS-CoV-2 polymerase. Nature Online ahead of print, doi:10.1038/s41586-020-2368-8 (2020).
- Ziebuhr, J. The Coronavirus Replicase. Curr Top Microbiol Immunol 287, 57-94, doi:10.1007/3-540-26765-4_3 (2005).
- Kirchdoerfer, R. N. & Ward, A. B. Structure of the SARS-CoV nsp12 polymerase bound to nsp7 and nsp8 co-factors. Nat Commun 10(1), 2342, doi:10.1038/s41467-019-10280-3 (2019).
- Wang, M. et al. Remdesivir and chloroquine effectively inhibit the recently emerged novel coronavirus (2019-nCoV) in vitro. Cell Res 30(3), 269-271, doi:10.1038/s41422-020-0282-0 (2020).
- Lehmann, K. C. et al. Discovery of an essential nucleotidylating activity associated with a newly delineated conserved domain in the RNA polymerase-containing protein of all nidoviruses. Nucleic Acids Res 43(17), 8416-8434, doi:10.1093/nar/gkv838 (2015).
- Cardone, G. et al. Maturation of the human Papillomavirus 16 capsid. mBio 5(4), e01104-01114, doi:10.1128/mBio.01104-14 (2014).


 Scarica il PDF dell'articolo
Scarica il PDF dell'articolo
Commenti