di Matteo Chiara1,2 & Graziano Pesole2,3
1 Dipartimento di Bioscienze, Università degli Studi di Milano
2 Istituto di Biomembrane Bioenergetica e Biotecnologie Molecolari, Consiglio Nazionale delle Ricerche (IBIOM-CNR)
3 Dipartimento di Bioscienze, Biotecnologie e Biofarmaceutica, Università degli Studi di Bari A. Moro
Indice dei contenuti
Ultimo aggiornamento: 17 novembre 2020
- Introduzione: dal DNA ai genomi (passando per un computer)
- Genomi e genomica per il controllo delle pandemie
- Genomica e SARS-CoV-2: risorse e applicazioni
- Condivisione delle informazioni: banche dati “primarie”
- Elaborazione delle informazioni: risorse “secondarie” [agg. 17/11/2020]
- Limiti e sfide per il futuro, oltre i genomi?
- Conclusioni
Per i non esperti
- Genoma
- RNA messaggeri
- Silencer (silenziatore)
- Enhancer (intensificatore)
- Promotore
- Organulo
- Metadato
Bibliografia
Introduzione: dal DNA ai genomi (passando per un computer)
La capacità di determinare in modo accurato il patrimonio genetico di un organismo, corrispondente alle informazioni presenti nel suo genoma, è stata una tappa fondamentale per lo sviluppo della moderna biologia molecolare1,2. La conoscenza della sequenza di un genoma è in grado di rivelare informazioni fondamentali sui meccanismi molecolari che regolano tutte le funzioni vitali. Per esempio, ci permette, tramite un processo chiamato annotazione, di identificare e caratterizzare i geni, ossia le porzioni del genoma che mediano e controllano i complessi processi metabolici alla base del funzionamento di tutti i tipi cellulari e quindi della vita stessa di un organismo.
Secondo la moderna teoria dell’evoluzione, tutti gli organismi viventi derivano da un unico antenato comune, tramite un processo di cambiamento (mutazione) e selezione che avrebbe avuto inizio oltre tre miliardi di anni fa3. Ne consegue che molti esseri viventi, e specialmente quelli più strettamente imparentati, condividono buona parte dei loro geni e mostrano alti livelli di similarità nella sequenza dei rispettivi genomi. Sfruttando questo principio, tramite il confronto diretto delle sequenze di uno o più genomi di organismi diversi, la disciplina nota come genomica comparata (confronto delle sequenze dei genomi) è in grado di ricavare informazione essenziali sulle leggi che governano l’evoluzione dei genomi e sulla localizzazione degli elementi di sequenza più importanti o indispensabili all’interno di un genoma (come i geni)4.
Lo stesso principio può essere applicato ed esteso anche ad altri ambiti della ricerca, come in genetica o in medicina, dove, per esempio, tramite il confronto tra sequenze genomiche di persone sane e persone affette da una malattia è possibile identificare varianti genetiche (porzioni del genoma più o meno piccole che differiscono a livello di sequenza) associate alla condizione patologica, ed eventualmente determinarne la funzione molecolare.
Le tecnologie di sequenziamento di nuova generazione
La genomica comparata e le varie applicazioni del sequenziamento del genoma hanno conosciuto un notevole sviluppo negli ultimi anni, dovuto alla disponibilità di un numero sempre crescente di sequenze complete di genomi. Questo ha prodotto una straordinaria rivoluzione nel campo delle Scienze della vita con l’avvento dell’era genomica. La causa di questa repentina trasformazione è da ricercarsi principalmente nello sviluppo di nuove metodologie di sequenziamento ad altissima capacità, meglio notte come tecnologie di sequenziamento nuova generazione o NGS (Next Generation Sequencing)5.
Le prime tecniche di laboratorio per la determinazione delle sequenze di acidi nucleici furono sviluppate alla fine degli anni ‘70 del Novecento nei laboratori di Frederick Sanger e Walter Gilbert (a entrambi è stato conferito il premio Nobel)1,2. Si trattava di tecniche molto laboriose e che potevano essere eseguite solo da personale altamente specializzato. Ovviamente la resa era molto limitata, se paragonata alle moderne tecniche di sequenziamento. Si stima, per esempio, che nei primi anni ‘80 applicando questi metodi di sequenziamento, una persona avrebbe potuto sequenziare un genoma di 30 000 paia di basi di DNA (circa le dimensioni del genoma del SARS-CoV-2) lavorando ininterrottamente per oltre 60 giorni.
Come accade per molti processi umani, le tecniche di sequenziamento sono state oggetto di costante ottimizzazione e miglioramento nel corso degli anni. Il crescente livello di automazione e l’ingegnerizzazione del processo di sequenziamento ha permesso di raggiungere livelli di produttività e accuratezza sempre maggiori. Per un resoconto dettagliato della storia delle diverse tecnologie di sequenziamento, dei rispettivi meccanismi di funzionamento e dei livelli di produttività raggiunti nel corso degli anni si rimanda al libro Fondamenti di Bioinformatica edito da Zanichelli6. In questo contesto, per rendere un’idea dei progressi raggiunti negli ultimi trentanni basti solo citare, che il progetto di sequenziamento del genoma umano (oltre 3 000 000 000 di paia di basi di DNA), così come ideato nel 1990 ha richiesto una spesa di tre miliardi di dollari per una durata di circa 15 anni. Al giorno d’oggi, applicando le più moderne tecnologie di sequenziamento, risultati paragonabili a quelli ottenuti dallo Progetto Genoma Umano, possono essere ottenuti in pochi giorni, con una spesa di circa 1000 dollari7 (Figura 1).
Figura 1 Costi del sequenziamento del genoma umano.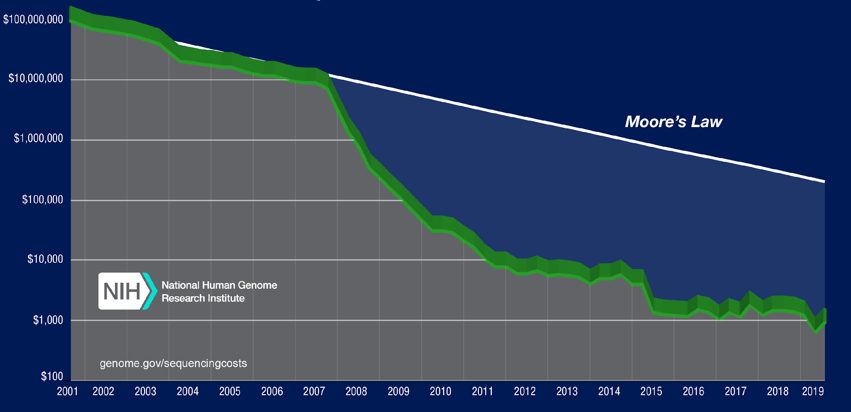
La figura mostra l’andamento dei costi stimati (asse Y, scala logaritmica) per il sequenziamento di un genoma umano in un lasso di tempo che comprende gli ultimi ventanni. La linea bianca (Moore’s Law) mostra l’andamento della prima legge di Moore, un postulato che descrive la diminuzione del prezzo dei componenti elettronici nello stesso periodo di tempo. Dal momento che la capacità dei componenti elettronici raddoppia (secondo la legge di Moore) ogni 18 mesi, nello stesso lasso di tempo il prezzo viene dimezzato (decrescita esponenziale). Ne consegue che la diminuzione dei costi di sequenziamento degli acidi nucleici negli ultimi anni ha conosciuto una riduzione più che esponenziale. [Fonte: NIH: genome.gov/sequencingcosts]
La ricostruzione della sequenza di un genoma è un processo complesso, che richiede la generazione, la gestione e l’analisi di una grande mole di dati. Questo concetto vale anche per genomi di dimensioni relativamente compatte, e fu subito evidente sin dal primo progetto per il sequenziamento di un genoma nei tardi anni ‘70. I ricercatori del gruppo di Frederick Sanger erano allora impegnati nella ricostruzione della sequenza del genoma del virus batteriofago ϕX1748. I dati di sequenziamento erano annotati nei quaderni di laboratorio di nove diversi ricercatori, ognuno impegnato ad analizzare un diverso segmento del genoma. La ricostruzione della sequenza completa richiedeva l’integrazione di tutti i dati di sequenziamento, problema che risultò da subito di non facile soluzione, dato che si trattava di unire in maniera coerente e codificata una grande mole di informazioni relative alla sequenza delle circa 5000 paia di basi che formano il genoma del virus. Fortunatamente, uno dei collaboratori di Sanger, Michael Smith aveva un cognato, Duncan McCallum, che lavorava come programmatore in un’azienda informatica di Cambridge. Per facilitare la ricostruzione della sequenza del genoma del fago, McCallum sviluppò un programma per computer (in linguaggio COBOL, su schede perforate), che consentiva di:
- integrare le sequenze prodotte dai diversi ricercatori;
- ricostruire la sequenza completa del genoma;
- cercare sequenze specifiche all’interno del genoma;
- tradurre in-silico le sequenze di DNA in proteina, grazie alla conoscenza del codice genetico.
Il programma si rivelò di grandissima utilità per la ricostruzione della sequenza del genoma del fago ϕX174 e dimostrò come l’uso dell’elaboratore e di programmi sviluppati ad hoc per l’analisi di sequenze biologiche fosse condizione irrinunciabile e necessaria per lo studio dei genomi. Nasceva proprio allora la disciplina oggi nota come bioinformatica.
Il successo dell’applicazione di metodi informatici per lo studio di sequenze biologiche fu tale, che nel corso di pochi anni, già nel 1981, si cominciarono a sviluppare banche dati dedicate per i dati di sequenziamento (EMBL9 e Genbank10, che tuttora costituiscono un punto di riferimento essenziale per i biologi di tutto il mondo) e strumenti appositamente studiati per l’interrogazione di banche dati di sequenze e la ricerca di sequenze simili, come i programmi FASTA11 e BLAST12, che sono a tutt’oggi tra gli strumenti più utilizzati dalla comunità scientifica mondiale. Nel corso degli anni, dato il costante aumento della quantità di dati prodotti, lo sviluppo di applicazioni software dedicate per la gestione delle analisi dei dati biologici di sequenziamento (ma non solo) è diventato sempre più centrale per la biologia, e la disciplina nota come bioinformatica è diventata fondamentale per ricostruire, analizzare e decodificare le sequenze dei genomi. Per una panoramica più completa delle molteplici applicazioni della bioinformatica si invita a fare riferimento a Fondamenti di Bioinformatica di Zanichelli6.
Come accennato in precedenza, il recente sviluppo della genomica (la scienza che studia i genomi) è legato a doppio filo allo sviluppo di nuove tecnologie di sequenziamento sempre più rapide ed efficienti. Negli ultimi 15 anni, queste tecnologie hanno rivoluzionato il modo di fare ricerca nell’ambito della biologia molecolare. Progetti di ricerca che prima avrebbero richiesto anni di sforzi e spese ingenti, sono ora realizzabili in tempi brevi e con l’investimento di risorse umane e somme relativamente modesti. Proprio per rimarcare la netta discontinuità con il passato è stato coniato l’acronimo NGS (Next Generation Sequencing) per qualificare queste nuove tecnologie. Le tecnologie di NGS permettono di svolgere più reazioni in parallelo, producendo così milioni di sequenze allo stesso tempo. L’aumento più che esponenziale della quantità di dati di sequenza generati da queste nuove tecnologie, ha richiesto un analogo incremento delle risorse computazionali necessarie per analizzare i dati, come anche lo sviluppo di nuovi algoritmi e software per rendere le analisi informatiche più trattabili ed efficienti. Questo processo ha reso la bioinformatica e le applicazioni della bioinformatica per l’analisi di dati di sequenziamento massivo sempre più centrali per la biologia.
Non esiste applicazione nell’ambito della biologia molecolare che non abbia tratto beneficio dall’avvento delle tecnologie di sequenziamento NGS: le applicazioni di queste tecnologie spaziano infatti dalla quantificazione degli mRNA in una cellula o in un tessuto, alla genetica di popolazione, alla ricostruzione di genomi, allo studio di fattori di trascrizione e dello stato della cromatina, e, più in generale, allo studio degli elementi regolatori dell’espressione genica (quali enhancer o silencer), o addirittura alla studio di intere comunità di microorganismi (metagenomica). Tutte queste analisi possono essere svolte in tempi brevi e a costi relativamente ridotti, producendo una mole di dati senza precedenti13.
L’assemblaggio di un genoma
Sebbene le nuove tecnologie di sequenziamento NGS siano state portatrici di enormi vantaggi, non tutti i problemi legati alla ricostruzione della sequenza di un genoma sono stati risolti. Una delle limitazioni principali è che, per quanto sofisticata, e salvo rarissime eccezioni, nessuna tecnologia di sequenziamento esistente è in grado di fornire una rappresentazione accurata, completa e univoca della sequenza del genoma di un organismo. Infatti, pur essendoci grandi differenze tra le tecnologie attualmente disponibili, nessuna di queste è in grado di leggere sequenze di DNA di lunghezza paragonabile a quella di un genoma di un organismo mediamente complesso. Si aggiunga inoltre, che a oggi, le tecnologie in grado di ricostruire le sequenze di maggiore lunghezza, tipicamente sono associate ad un alto tasso di errore5.
Proprio per questo motivo, così come accadeva negli anni ‘70, la ricostruzione della sequenza completa del genoma richiede l’utilizzo di programmi e strumenti software dedicati, per l’analisi dei dati di sequenziamento, tramite un processo noto come assemblaggio14.
Con il termine assemblaggio di un genoma si intende l’operazione tramite la quale i dati ottenuti da un progetto di sequenziamento genomico (che rappresentano porzioni discrete del genoma) vengono concatenati formando sequenze contigue di dimensioni maggiori, idealmente corrispondenti a ciascuna delle molecole (cromosomi, genomi di organuli e plasmidi) di DNA presenti nella cellula. Le sequenze ottenute in questo modo vengono chiamate contigui, o contig in inglese. Il processo di assemblaggio è solitamente effettuato da appositi programmi per computer, detti assemblatori. Il risultato ottimale è costituito da una serie di contig completi che rappresentino in un rapporto di 1:1 ciascuna delle molecole presenti nel genoma. Purtroppo, dal momento che la maggior parte dei genomi degli organismi complessi contiene lunghe sequenze ripetute, che causano problemi nell’assemblaggio, questo risultato è particolarmente difficile da ottenere.
L’annotazione genomica
Una volta ricostruita la sequenza di un genoma, il passaggio successivo consiste nella determinazione dei suoi elementi funzionali, quali i geni, i promotori (gli elementi che regolano l’utilizzo dei geni), e le altre sequenze regolatorie (enhancer, silencer). Il processo tramite il quale i diversi elementi funzionali di un genoma vengono riconosciuti viene detto annotazione. L’annotazione di un genoma viene solitamente eseguita utilizzando appositi strumenti bioinformatici, e/o producendo nuovi dati di sequenziamento mirati per l’annotazione dei geni (per esempio, il sequenziamento di prodotti della trascrizione del genoma, costituiti da più RNA discreti)15.
L’annotazione basata puramente sull’utilizzo di strumenti software viene detta annotazione in silico. La genomica comparata è una delle più efficaci strategie per l’annotazione in silico dei genomi, infatti, quando possibile, il confronto con una sequenza genomica relativamente simile e già “annotata” consente una rapida identificazione degli elementi funzionali conservati (omologhi). In alternativa gli elementi funzionali di un genoma possono anche essere “predetti” applicando metodi ab initio. Questi metodi sfruttano algoritmi probabilistici per determinare, date le proprietà di una sequenza nucleotidica (come la composizione o la presenza di particolari motivi di sequenza), quanto sia probabile che essa contenga un gene o un altro elemento funzionale. Le moderne tecnologie di sequenziamento consentono inoltre di applicare particolari protocolli di laboratorio che permettono di sequenziare i prodotti intermedi degli elementi funzionali di un genoma, ossia i trascritti. Per esempio, è possibile sequenziare in un determinato tipo cellulare gli RNA messaggeri o piccoli RNA regolatori (sncRNA, short non coding RNA) e ottenere informazioni sulle regioni che li codificano semplicemente “mappando” (cercando le regioni genomiche con sequenza identica) le sequenze ottenute sul genoma di riferimento dello stesso organismo.
Tutti questi approcci, naturalmente, possono essere combinati per ottenere un’annotazione più completa.
Banche dati genetiche
Una volta che una sequenza genomica è stata ricostruita e annotata, questa viene solitamente resa disponibile alla comunità scientifica tramite apposite risorse e banche dati, che consentono tra le altre cose la navigazione tra le annotazioni, il rapido confronto con altre sequenze già disponibili, l’estrazione di una o più sequenze simili, e la visualizzazione in forma grafica degli elementi genomici annotati. Tra gli esempi più noti possiamo citare Genbank, una banca dati di sequenze biologiche curata dall’NIH (National Institute of Health)10, una delle più rinomate istituzioni pubbliche per la ricerca medica a livello mondiale, oppure la banca dati di sequenze ENA16, sviluppata e mantenuta da ELIXIR, l’infrastruttura europea per le Scienze della vita, o ancora i genome browser di UCSC17 ed ENSEMBL18: degli strumenti software avanzati che consentono la navigazione attraverso gli elementi funzionali annotati in un genoma tramite una intuitiva e semplice interfaccia grafica.
La disponibilità pubblica e gratuita dei dati per tutta la comunità scientifica, unita ai vari strumenti che le banche dati specializzate mettono a disposizione dei ricercatori, è di fondamentale importanza per il continuo miglioramento della nostra conoscenza dei genomi e dei meccanismi che ne governano l’evoluzione e l’utilizzo. Lo sviluppo e il miglioramento di queste risorse e infrastrutture è pertanto oggetto di continuo ed incessante sforzo da parte della comunità mondiale dei bioinformatici.
Genomi e genomica per il controllo delle pandemie
Tra la fine del 2013 e l’inizio del 2014, una febbre emorragica letale si è diffusa, non diagnosticata per mesi, in tutta la Guinea meridionale (Guinée forestière). Quando la malattia è stata diagnosticata come ebola, il virus si era diffuso in tre Paesi19, probabilmente oltre il punto in cui le misure di contenimento a livello locale, come l’isolamento e il controllo mirato delle catene di infezione, avrebbero potuto contenere l’epidemia nascente. Nel 2015, una nuova malattia simile alla febbre dengue è stata implicata in un drammatico aumento dei casi di microcefalia in Brasile; un anno dopo, le analisi hanno rivelato che il virus zika si era diffuso nelle Americhe, inosservato dai sistemi di sorveglianza esistenti, dalla fine del 201320.
Queste recenti esperienze dimostrano come, sebbene i sistemi di sorveglianza della salute pubblica si siano evoluti per soddisfare le mutevoli esigenze della popolazione mondiale, continuiamo a sottovalutare drasticamente la nostra vulnerabilità agli agenti patogeni, sia vecchi sia nuovi21. I recenti eventi in Africa occidentale e Brasile evidenziano lacune nei sistemi di sorveglianza delle malattie infettive, in particolare quando si tratta di individuare nuovi patogeni o agenti patogeni la cui area geografica si è estesa a una nuova regione. Nonostante le lezioni apprese da epidemie precedenti22, come l’epidemia di sindrome respiratoria acuta grave (SARS) nel 2002–2003 e la pandemia influenzale del 2009, le minacce infettive continuano a sorprendere e, a volte, a sopraffare i sistemi di controllo a livello locale e globale.
L’implementazione di sistemi di monitoraggio di agenti patogeni altamente infettivi e potenzialmente letali richiede metodi di sorveglianza tempestivi e completi. I sistemi di monitoraggio attualmente usati si basano sul conteggio dei casi a livello locale e su semplici tecniche di sequenziamento parziale (genotipizzazione) per la classificazione dei patogeni; questi metodi di sorveglianza potrebbero essere notevolmente migliorati attraverso i più moderni strumenti della genomica.
Il sequenziamento completo del genoma di agenti patogeni è stato utilizzato per decenni per comprendere la trasmissione nelle epidemie virali, dai primi studi sull’hantavirus negli Stati Uniti23, agli studi sul virus dell’immunodeficienza umana (HIV) nel Regno Unito24; più recentemente, l’approccio è stato esteso con successo anche ai patogeni batterici25. L’epidemiologia genomica, la disciplina che utilizza l’approccio del sequenziamento del genoma di un patogeno per lo studio di eventi epidemici, consente in primo luogo di riconoscere l’agente patogeno, e successivamente di determinare allo stesso tempo, tramite il confronto dei genomi, sia i meccanismi che governano l’evoluzione del genoma del patogeno stesso, sia singoli eventi di trasmissione individuale all’interno dei focolai epidemici (catene di contagio)25. Più di recente, questo approccio si è dimostrato uno strumento cruciale nella nostra risposta in tempo reale ai nuovi focolai di malattie infettive26,27.
Molti patogeni, e specialmente i virus, mutano la sequenza del proprio genoma molto rapidamente, con la conseguenza che, patogeni isolati anche da casi di infezione strettamente collegat,i mostrano differenze significative a livello della sequenza genomica, e sono chiaramente distinguibili. Queste differenze possono essere usate per trarre conclusioni epidemiologiche e ricostruire probabili catene di contagio28. Il sequenziamento del genoma completo di un virus emergente può fornire una notevole quantità di informazioni sulla natura del patogeno, attraverso il confronto con le sequenze esistenti. Man mano che un numero maggiore di sequenze diventa disponibile, l’analisi della diversità genetica della popolazione del nuovo patogeno può fornire stime della velocità di diffusione della malattia e aiutare a prevederne il decorso futuro.
Nelle indagini per lo studio delle catene di trasmissione, le varianti genetiche vengono usateper identificare eventi di trasmissione da persona a persona, tramite l’analisi delle varianti genetiche condivise tra i patogeni isolati dai diversi casi di un focolaio. In questo modo è possibile ricostruire in modo dettagliato l’intera rete di trasmissione28.
Le indagini genomiche di eventi di diffusione di un patogeno su larga scala, come le epidemie, sono molto diverse: data l’alta diffusione della malattia solo un sottoinsieme dei genomi del patogeno, può essere isolato e sequenziato. Pertanto, l’obiettivo è usare i dati di sequenziamento genomico per comprendere le dinamiche generali dell’epidemia e i possibili meccanismi di evoluzione del patogeno. Questo approccio che integra principi di epidemiologia e biologia evoluzionistica viene definito filodinamica29. Dal momento che la maggior parte dei patogeni, e in particolare virus a DNA a filamento singolo, virus a RNA e molte specie batteriche, mutano in maniera molto rapida, tramite la filodinamica è possibile combinare osservazioni epidemiologiche con analisi evolutive, per determinare come le dinamiche che governano l’evoluzione del genoma del patogeno possano influire sulle manifestazioni cliniche della malattia30.
Riassumendo, si possono descrivere almeno tre approcci genomici per l’epidemiologia:
- la sequenza del genoma di un patogeno emergente può essere determinata tramite le moderne tecnologie di sequenziamento;
- la disponibilità delle sequenze del genoma, isolate da persone contagiate, permette di ricostruire la catena di contagio;
- la disponibilità di un numero sempre crescente di genomi consente alla disciplina nota come filodinamica di ricostruire con precisione gli eventi di trasmissione e l’evoluzione del patogeno su scala più ampia.
Genomica e SARS-CoV-2: risorse e applicazioni
La recente applicazione della genomica per il monitoraggio della pandemia della COVID-19 ha evidenziato l’utilità degli approcci basati sul sequenziamento del genoma di un patogeno nella risposta in tempo (quasi) reale a una gravissima crisi di salute pubblica.
A cavallo tra il 2019 e il 2020, un gruppo di scienziati, ha applicato una particolare tecnica di sequenziamento, che consente di determinare la sequenza dell’RNA di tutte le specie viventi presenti in un campione biologico/ambientale (sequenziamento metagenomico del RNA totale) per analizzare un campione di fluido di lavaggio broncoalveolare (BALF) ottenuto da un singolo paziente a Wuhan, in Cina, dove erano stati segnalati diversi casi di gravi infezioni respiratorie31. Tramite l’applicazione di strumenti bioinformatici, Wu e colleghi sono riusciti a ricostruire dai dati di sequenziamento il genoma del potenziale agente patogeno, un nuovo coronavirus successivamente denominato SARS-CoV-2. Nei primi giorni del gennaio 2020, la sequenza completa del genoma virale è stata depositata in banca dati e resa disponibile all’intera comunità scientifica. La ricostruzione della sequenza del genoma del nuovo agente patogeno ha facilitato lo sviluppo di test diagnostici molecolari rapidi in tutto il mondo32. Questi test sono stati fondamentali per monitorare e arginare il diffondersi della malattia in tempo reale. Purtroppo, come noto, il SARS-CoV-2, il virus che causa la pandemia di COVID-19 attualmente in corso, si è diffuso rapidamente a livello globale. Ancora una volta le tecniche di analisi basate sulla ricostruzione della sequenza del genoma del patogeno sono state il perno di uno sforzo globale e coordinato per studiare la trasmissione della malattia e l’evoluzione virale sia a livello nazionale che internazionale.
A titolo di esempio, basti citare il progetto SPHERES33 (SARS-CoV-2 Sequencing for Public Health Emergency Response, Epidemiology and Surveillance) negli Stati Uniti o il progetto COG-UK34 (COVID-19 Genomics UK) nel Regno Unito. Quest’ultimo, lanciato nel marzo 2020, è probabilmente il modello di sistema di monitoraggio della pandemia di COVID-19 più completo e accurato a livello mondiale. Il COG-UK consiste in una capillare rete di sorveglianza genomica a livello nazionale che mira a tracciare la trasmissione di SARS-CoV-2, identificare nuove mutazioni nel genoma e integrare i dati genomici con i dati epidemiologici e sanitari. A oggi (14 agosto 2020), il COG-UK ha determinato la sequenza di più di 35 000 genomi di SARS-CoV-2.
Questi dati sono stati cruciali per ricostruire in maniera accurata le dinamiche di trasmissione della COVID-19 nel Regno Unito. Sulla scorta di SPHERES e COG-UK, iniziative simili sono state intraprese a livello nazionale in vari paesi colpiti dalla COVID-1935,36,37,38. La disponibilità di questa grandissima quantità di dati genomici in tempo reale è risultata fondamentale per tracciare le catene di contagio e per opporre una resistenza informata ed organizzata alla diffusione del SARS-CoV-2.
Condivisione delle informazioni: banche dati “primarie”
Il progetto GISAID (Global Initiative on Sharing All Influenza Data) è oggi il punto di riferimento a livello mondiale per la condivisione dei dati genomici di patogeni virali e lo studio di mutazioni emergenti che possono essere rilevanti per il controllo delle epidemie e lo sviluppo di vaccini39. L’iniziativa che ha portato alla nascita di GISAID affonda le sue radici nel 2006, quando la riluttanza alla condivisione dei genomi del virus dell’influenza aviaria H5N1, ha creato un’emergenza globale, mettendo a nudo alcune gravi carenze e limitazioni nel sistema di sorveglianza GINS (Global Influenza Surveillance Network) dell’Organizzazione Mondiale della Sanità (OMS)40. Sulla scorta dell’esperienza acquisita, la comunità scientifica ha elaborato nuovi principi e regole per promuovere la condivisione internazionale di tutti i dati sui virus influenzali e rendere pubblici i risultati in maniera collaborativa. Ciò che è iniziato come un’espressione di intenti, si è dimostrato nel tempo un meccanismo indispensabile per favorire, tramite la condivisione dei dati, una reazione più coordinata ed efficiente alle epidemie influenzali.
La banca dati EpiFlu, la risorsa di GISAID per la condivisione dei dati dei genomi di virus influenzali, è stata di fondamentale importanza nella risposta alla pandemia di influenza A (H1N1) del 2009, consentendo di monitorare prontamente l’evoluzione del nuovo virus mentre si diffondeva a livello globale41. Successivamente, nel 2013, la disponibilità di sequenze genomiche del virus H7N9 è stato fondamentale per generare, sviluppare e testare vaccini, mediante approcci di biologia sintetica, entro poche settimane dall’isolamento del virus stesso42.
Al momento, il portale EpiCov, la risorsa di GISAID sviluppata appositamente per raccogliere i dati della pandemia di COVID-19, èl’archivio più usato e completo di dati genomici del SARS-CoV-2. Il sito EpiCoV fornisce una raccolta di oltre 80 000 genomi completi, isolati da oltre 80 Paesi (dati raccolti il 14 agosto 2020). Oltre ai genomi, il portale EpiCoV rende disponibili diversi metadati, anche se limitati, incluso il tipo di campione da cui il virus è stato caratterizzato, la tecnologia di sequenziamento e i protocolli applicati, ma anche annotazioni cliniche di base, ovvero lo stato del paziente (per esempio, ricoverato in ospedale o asintomatico), il sesso e l’età. Oltre ai dati stessi, l’EpiCoV rende disponibili, su base settimanale, elaborazioni dei dati genomici per delineare le mutazioni più ricorrenti e diffuse nel genoma del virus, e possibili mutazioni con potenziale impatto per la clinica o per la diagnostica. Sebbene i dati in EpiCov siano pubblicamente accessibili, l’accesso è riservato solo agli utenti registrati, i dati non possono essere redistribuiti e il loro uso è limitato ai soli scopi di ricerca. Infine, i dati di sequenziamento non elaborati (le sequenze non assemblate) non possono essere depositati.
Per questi motivi, la Research Data Alliance (RDA)43 un gruppo di scienziati che si occupa di definire i principi ottimali per la condivisione dei dati scientifici, ha elaborato una serie di raccomandazioni e linee guida, per facilitare una condivisione ancora maggiore dei dati di ottenuti dal sequenziamento di SARS-CoV-2. In particolare, l’RDA consiglia fortemente che oltre a essere resi disponibili attraverso risorse ad accesso controllato, come GISAID, i dati dei genomi del SARS-CoV-2, ma anche i dati primari di sequenziamento, come ottenuti direttamente dall’apparato di sequenziamento, dovrebbero essere depositati anche in banche dati ad accesso libero, come il portale per i genomi virali sviluppato da Genbank10 o l’equivalente portale per la raccolta dei genomi di SARS-CoV-2 sviluppato dalla banca dati europea ENA16. Il libero accesso ai dati di sequenziamento primari ancora non elaborati è fondamentale per garantire la completa trasparenza e la riproducibilità delle analisi effettuate dai diversi gruppi di ricerca. Inoltre, la loro aggregazione può consentire, nell’immediato o nel futuro, l’esecuzione di nuove analisi, che tramite l’applicazione di strumenti bioinformatici migliori o diversi possono ricavare nuove informazioni sui meccanismi di evoluzione del virus o sulla patogenesi della malattia.
Elaborazione delle informazioni: risorse “secondarie”
In risposta alla pandemia di COVID-19, sono state generate quantità di dati che non trovano precedenti nella storia della scienza moderna. Navigare tra questa enorme mole di dati per trovare le informazioni di proprio interesse può essere un processo non sempre agevole e immediato. Per questo motivo la comunità scientifica si è rapidamente attivata per implementare infrastrutture computazionali per facilitare l’accesso e il recupero di diversi tipi di dati associati alla COVID-19.
Il portale COVID-19 dell’Istituto di Bioinformatica Europeo (EBI, European Bioinformatics Institute) e il portale gemello sviluppato da NIH sono probabilmente le risorse che al momento attuale forniscono il catalogo più completo per navigare, accedere e ottenere i dati di sequenziamento di SARS-CoV-2 e gli strumenti bioinformatici migliori da utilizzare per la loro analisi. Il portale protocols.io colleziona una raccolta di oltre 150 protocolli di laboratorio e in silico (bioinformatici) per la generazione, la manipolazione, l’analisi e la deposizione in banche dati pubbliche di dati SARS-CoV-2. Allo stesso modo, un elenco dettagliato di protocolli di laboratorio, metodi bioinformatici e banche dati che contengono dati di sequenziamento di SARS-CoV-2 è stato reso disponibile, tramite la risorsa Github, dal Centro Statunitense per il Controllo e la prevenzione delle malattie (CDC). Iniziative simili non mancano anche a livello nazionale, come il portale per l’accesso ai dati sviluppato da COG-UK o il COVID-19 Data Portal Sweden, sviluppato appunto dai ricercatori svedesi.
Oltre ai portali per la condivisione dei dati, la comunità scientifica ha sviluppato diversi strumenti software e strategie di analisi per superare la sfida della loro integrazione e interpretazione, per esempio sono state sviluppate applicazioni e metodi di facile uso per la classificazione dei ceppi di SARS-CoV-2, basati sia sul confronto diretto di genomi completi tramite alberi filogenetici sia su strategie più stratificazione più robuste che usano solamente le varianti genomiche più frequenti44,45,46. Per facilitare l’interpretazione di questi risultati, sono inoltre stati sviluppati strumenti web dedicati per visualizzare in modo interattivo i ceppi virali circolanti in diverse aree del pianeta (per esempio, Microreact e Nextstrain47,48; Figura 2).
Figura 2 Esempio di sistemi per la visualizzazione di dati filogeografici.
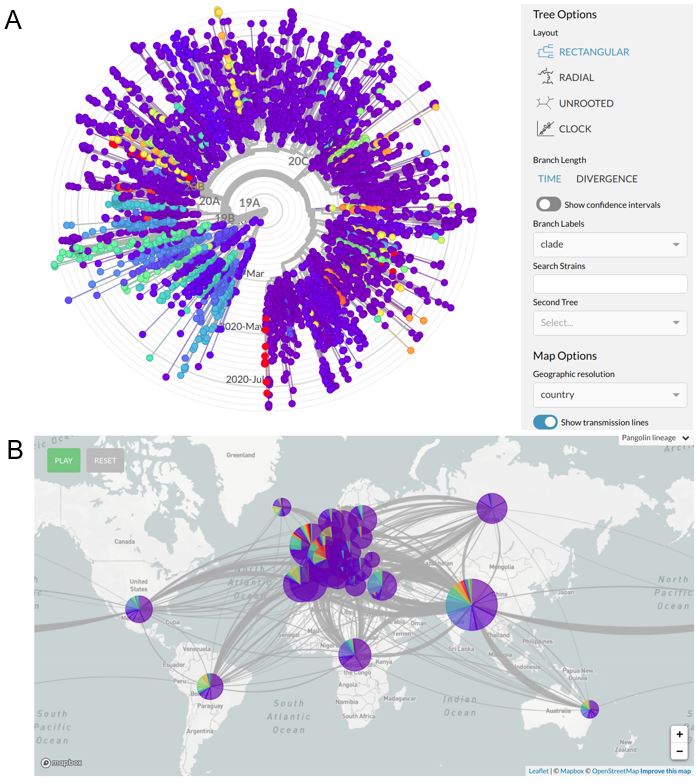
A Un albero filogenetico, un tipo di rappresentazione grafica delle distanze genetiche tra organismi che consente di identificare in maniera visiva le entita tra loro più simili. Nella piattaforma Nexstrain gli alberi filogenetici possono essere usati per visualizzare i rapporti di parentela tra gli isolati di SARS-CoV-2. In questo caso i colori indicano i diversi tipi virali. I menù a tendina consentono vari livelli di configurazione. B La mappa dettagliata delle connessioni tra i diversi ceppi circolanti nel mondo consente di ottenere una rappresentazione in tempo quasi reale delle dinamiche di trasmissione del virus. [Schermate tratte dal sito nextstrain.org]
Sfruttando le sequenze genomiche disponibili in GISAID, Korber e colleghi49 hanno sviluppato una serie di strumenti bioinformatici ad hoc per tenere traccia dei cambiamenti nella glicoproteina spike di SARS-CoV-2, che media l’ingresso nella cellula ospite e che costituisce uno dei migliori target per lo sviluppo di un vaccino. Integrando le analisi genomiche con i dati clinici, gli autori hanno individuato una particolare mutazione amminoacidica nella sequenza della proteina spike (D614G) che sembrerebbe correlata a cariche virali potenzialmente più elevate nei pazienti e potrebbe essere quindi il risultato di un processo di adattamento di SARS-CoV-2 all’ospite umano. Al momento attuale le implicazioni cliniche di questa osservazione, che in mancanza di ulteriori validazioni di laboratorio costituisce solo una valida teoria, non sono ancora chiare dato che, anche a giudizio degli stessi autori, la presenza di questa variante non sembra essere associata a una maggiore gravità della malattia. Osservazioni simili, ancora in attesa di validazione, sono state avanzate anche da altri autori, e costituiscono un valido punto di partenza per l’esecuzione di analisi più mirate per la comprensione dei meccanismi molecolari alla base della patogenesi di COVID-19.
[Agg. 17/11/2020] È stato recentemente stabilito che, in realtà, la variante D614G è effettivamente in grado infettare le cellule del tratto respiratorio superiore e dei polmoni in maniera più efficiente. La variante G614 replica di più nelle cellule del tratto respiratorio superiore ed è causa di cariche virali più elevate, ma non nei polmoni64. Per tale motivo, questa variante non correla con le forme più severe di malattia. Nelle cavie infettate precedentemente con la variante D614, la risposta immunitaria contro la variante G614 è stata molto più forte. Questo significa che i vaccini giunti alla fase 3 della sperimentazione sono ancora l’obbiettivo da raggiungere per uscire dalla pandemia. Tuttavia, la capacità di riconoscere questi antigeni da parte degli anticorpi monoclonali, un’altra grande speranza terapeutica per la COVID-19, deve essere testata di nuovo con cura. Infatti, tutti gli anticorpi sintetici creati finora sono stati progettati tenendo in considerazione il tipo D614. La glicina (G) nella posizione 614 di Spike sembra far cambiare conformazione alla proteina, limitando il riconoscimento da parte di alcuni anticorpi64. [fine agg.]
Limiti e sfide per il futuro, oltre i genomi?
La maggior parte dei sistemi di sorveglianza degli agenti patogeni integra diversi tipi di dati, ottenuti, incluso quelli genomici, per effettuare la sorveglianza di una specifica malattia. La condizione patologica di interesse viene monitorata a diversi livelli, tramite test diagnostici, modelli epidemiologici, dati genomici, o in caso di focolai emergenti, rapide notifiche di situazioni anomale. Nonostante i notevoli progressi raggiunti nei decenni precedenti, più gruppi di esperti hanno ripetutamente sottolineato la necessità di una migliore capacità di sorveglianza delle malattie infettive50. Idealmente, un sistema di sorveglianza globale dovrebbe essere basato su un approccio più agnostico e non specifico per un particolare patogeno51. Un sistema di sorveglianza di questo tipo dovrebbe sfruttare e integrare nuovi tipi di dati, come i big data che ci vengono messi a disposizione dalle nuove tecnologie digitali. Per esempio, tramite il monitoraggio di diversi flussi di dati, quali l’assenteismo scolastico o dei dipendenti sul luogo di lavoro, l’acquisto di particolari generi alimentari o articoli specifici in farmacia, o il flusso di pazienti nelle strutture ospedaliere, o ancora l’analisi automatizzata di parole o frasi di tendenza sui social media, come Twitter52 o nelle ricerche su Internet53.
Questo nuovo approccio di sorveglianza, noto come epidemiologia digitale, potrebbe essere molto utile per il monitoraggio digitale, in tempo reale, delle malattie infettive54. Nell’epidemiologia digitale, le informazioni vengono prima recuperate da una serie di fonti, inclusi i media digitali, i notiziari, i rapporti ufficiali e il crowdsourcing; successivamente, i dati rilevanti per l’epidemiologia vengono estratti ed elaborati in una serie di rapporti, che vengono poi analizzati per individuare possibili situazioni critiche. Al momento, sono operative nel mondo almeno 50 diverse piattaforme per l’epidemiologia digitale54, benché queste non siano ancora integrate in sistemi di monitoraggio ufficiale riconosciuti dalla comunità scientifica, la loro natura flessibile e la possibilità di ottenere dati in tempo reale rendono queste piattaforme efficaci strumenti per raccogliere informazioni su un’epidemia, in particolare nei contesti privi di sistemi tradizionali di sorveglianza delle malattie.
Recenti esperienze con le epidemie di ebola e zika, hanno dimostrato come i domini dell’epidemiologia digitale e la genomica potrebbero essere in futuro integrati per offrire un quadro più dettagliato e chiaro di una epidemia in corso. Durante il monitoraggio dell’epidemia di ebola in Africa occidentale, gli approcci basati sull’epidemiologia digitale hanno rilevato come le zone di interfaccia tra popolazioni rurali e popolazioni urbane costituissero uno degli snodi privilegiati per la diffusione del virus55, facendo eco ai risultati ottenuti tramite uno studio genomico delle principali catene di contagio19. Durante l’epidemia di zika in Brasile, Majumder e colleghi hanno usato Google Trends per stimare il numero riproduttivo di base R0 del virus56, ottenendo una stima del tutto analoga a quella ottenuta tramite analisi filodinamiche dei dati genomici (1,29–3,85)20. Questi risultati indicano che entrambi i tipi di approccio, filodinamica ed epidemiologia digitale, possono essere utilizzati per il calcolo di parametri epidemiologici fondamentali che aiutano a comprendere la diffusione del virus e ad organizzare un’adeguata risposta per la tutela della salute pubblica.
In un futuro non lontano non è difficile immaginare un’integrazione completa dei dati genomici con altri tipi di dati epidemiologici digitali57. Il potenziale di un sistema di sorveglianza epidemiologico di questo tipo sarebbe molto maggiore rispetto a quello dei sistemi attualmente in uso. I dati epidemiologici digitali potrebbero essere usati per indirizzare i ricercatori verso regioni del mondo in cui si stanno verificando possibili situazioni critiche dal punto di vista epidemiologico. Successivamente, in queste regioni si potrebbero intraprendere iniziative di sequenziamento sistematico di campioni raccolti da specie selvatiche infette, o da pazienti umani, per identificare possibili nuovi agenti patogeni. I dati di sequenziamento del genoma potrebbero a loro volta essere integrati in tempo reale con metadati sempre più ricchi, e venire resi disponibili tramite piattaforme web dedicate, come Microreact per l’analisi collaborativa dei dati di sequenziamento e Nextstrain per la visualizzazione dei risultati. Queste risorse o risorse analoghe, già usati nelle risposte delle epidemie di ebola e zika o nella risposta alla pandemia di COVID-19 attualmente in corso, fungerebbero a loro volta da punto di aggregazione per una rete globale esperti che potrebbero contribuire alle analisi filodinamiche ed epidemiologiche, consentendo l’identificazione di possibili eventi di spillover, l’espansione della popolazione di un patogeno e/o la trasmissione di un nuovo agente infettante da persona a persona. I risultati sarebbero immediatamente condivisi con il personale in prima linea per nella lotta alle malattie infettive, epidemiologi, veterinari e operatori sanitari, consentendo di attuare interventi di contenimento rapidi e mirati per mitigare l’ulteriore diffusione.
Conclusioni
L’adattamento di un nuovo patogeno alla specie umana, i cosiddetti eventi di salto di specie o spillover, non è un fenomeno del tutto nuovo nella storia dell’umanità, anche se fortunatamente eventi della portata della pandemia di COVID-19 costituiscono un unicum a memoria umana58. La possibilità di condividere in tempo reale sempre più informazioni, di analizzare i dati tramite algoritmi sempre migliori e più efficienti e di ottenere risposte rapide e complete a problemi complessi in maniera tempestiva è sicuramente una delle armi principali che in un futuro non lontano consentirà all’umanità di approntare reti sempre più capillari ed efficienti per controllare e circoscrivere possibili situazioni di emergenza, come eventuali epidemie causate dall’insorgenza di nuovi agenti patogeni59. È facile prevedere che la bioinformatica e la genomica giocheranno un ruolo sempre più centrale nell’implementazione e nella messa in funzione di tali sistemi di monitoraggio. Come dimostrato anche dalla pronta ed efficiente risposta che queste discipline hanno saputo mettere in campo per contrastare l’attuale situazione di emergenza.
La portata degli sforzi messi in campo per la produzione e l’analisi dei dati genomici SARS-CoV-2 non ha precedenti nella storia della scienza moderna, infatti la produzione di tale mole di dati in un lasso di tempo così ridotto sarebbe stata inconcepibile solo pochi anni fa. Tuttavia, per elaborare strategie sempre migliori, in futuro il processo decisionale dovrà essere sempre più rapido e informato con dati accurati e in tempo reale e, idealmente, integrare diversi livelli di informazione, inclusi dati molecolari e genetici sia del patogeno che dell’ospite, dati epidemiologici e demografici, e dove possibile lo stato di salute e la storia clinica dei pazienti.
Per quanto la risposta della comunità scientifica alla pandemia di COVID-19 sia stata incredibilmente rapida ed efficiente, si registrano ancora alcune criticità che dovranno sicuramente essere risolte in un futuro non lontano.
A questo proposito è chiaro che la standardizzazione dei protocolli e dei metodi usati per produrre e analizzare i dati, e lo sviluppo di risorse integrate per consentire un accesso rapido e senza soluzione di continuità a tutte le informazioni disponibili, costituisce una delle sfide più rilevanti per il futuro della biologia moderna. Infatti, mentre sono attualmente disponibili numerose banche dati e risorse per fornire l’accesso ai dati di sequenziamento del SARS-CoV-2, l’integrazione di tutti i dati e metadati incorporati in questi archivi non è sempre semplice e limita possibili applicazioni e studi futuri. Per esempio, secondo le nostre analisi, a causa di incongruenze nei sistemi di deposizioni dei dati/o errori nelle annotazioni, tutte le principali banche dati di sequenze genomiche di SARS-CoV-2 mostrano un certo grado di ridondanza, e contengono informazioni equivalenti e sovrapposte che non sono facilmente discernibili. In assenza di regole chiare e ben definite per la deposizione dei dati e l’annotazione dei metadati, è quasi impossibile capire con precisione, e in quale misura, le informazioni e i dati vengono duplicati in diversi archivi. Questo fenomeno complica l’integrazione dei dati disponibili e ha profonde implicazioni anche per l’esecuzione di studi di associazione statistica che potrebbero consentire l’identificazione di varianti genetiche nell’ospite o nel virus, eventualmente associate a una forma più grave della malattia. Inoltre, benché la quasi totalità dei genomi di SARS-CoV-2 attualmente disponibili provenga da istituti clinici, i dati dei pazienti spesso vengono non vengono messi a disposizione della comunità scientifica, nemmeno in forma aggregata. Questo limita sensibilmente il tipo di analisi che è possibile eseguire. Considerazioni simili si applicano per le sperimentazioni cliniche per la valutazione dell’efficacia dei vaccini e di altri approcci terapeutici.
Gli studi di associazione genetica60 si sono dimostrati fondamentali in diversi ambiti per capire più a fondo le caratteristiche del nostro genoma. È ragionevole ipotizzare che se fossero disponibili campioni statistici di dimensioni adeguate, tali studi potrebbero essere ugualmente utili per comprendere al meglio la fisiologia del SARS‐CoV‐2 e le diverse manifestazioni cliniche della COVID‐19, con possibili applicazioni anche nella progettazione di nuovi vaccini e approcci terapeutici. Tuttavia, nonostante, alcune iniziative degne di nota61,62,63, attualmente, l’esecuzione di studi di associazione su larga scala per la COVID-19 è limitata, probabilmente anche a causa delle difficoltà nell’ottenere e integrare tutti i dati e metadati richiesti.
Per quanto dal punto di vista tecnico, tecnologico e scientifico l’umanità abbia compiuto progressi notevoli, la pandemia di COVID-19 ha sicuramente evidenziato delle criticità preoccupanti, anche e specialmente nei sistemi di comunicazione e gestione delle informazioni64. Infatti, e talvolta anche nel nostro Paese, notizie o nozioni incomplete, imprecise o semplicemente false hanno circolato con troppa facilità su diversi canali di comunicazione, esponendo la popolazione a paure e rischi del tutto immotivati. Per questo auspichiamo che in futuro, i sistemi di monitoraggio delle pandemie possano essere integrati, non solo per incorporare una quantità sempre maggiore di dati, ma anche per consentire una più corretta e accurata comunicazione delle informazioni rilevanti alla popolazione. Ovviamente un tale processo richiede anche una piccola rivoluzione culturale, per un più corretto uso dei mezzi di comunicazione e dei social media, e passa inevitabilmente dalla promozione di una più seria e diffusa cultura scientifica nella popolazione (ma anche nei responsabili dei media).
Un processo, che grazie ai moderni mezzi di comunicazione, potrebbe articolarsi anche attraverso iniziative più capillari e inclusive di citizen science, con la partecipazione diretta dei cittadini a grandi progetti di ricerca (per esempio, attraverso l’uso di app su cellulari che comunichino informazioni critiche per il monitoraggio di episodi pandemici, come l’insorgenza di febbre o di altri sintomi). Attraverso questo nuovo paradigma, che coinvolge più direttamente i cittadini in progetti di ricerca scientifica, per esempio rendendoli parte attiva nella raccolta, nel processamento e in alcune fasi delle analisi dei dati, sarebbe possibile infatti rendere la cittadinanza più edotta nel corretto utilizzo del metodo scientifico, riducendo il dilagare di notizie infondate, negazionismi e altre amenità di vario tipo che sono spesso veicolate in maniera non corretta attraverso i social media.
Per i non esperti
Genoma
Gli acidi nucleici, macromolecole biologiche costituite da lunghe catene di nucleotidi, sono i componenti chimici dei genomi, e possono essere di due tipi diversi (DNA o RNA), a seconda che lo zucchero presente nei nucleotidi sia il ribosio (RNA) o il deossiribosio (DNA).
L’informazione biologica, ovvero il codice universale della vita, è racchiusa nelle sequenze dei nucleotidi, che possono essere di quattro tipi diversi: adenina, citosina, timina (uracile nell’RNA) e guanina (A, C, T o U, G).
La maggior parte dei genomi è composta di DNA, tipicamente nella forma di due filamenti complementari (A e G si appaiano rispettivamente con T/U e C, T nel DNA e U nell’RNA), ma alcuni genomi virali possono essere costituiti da RNA.
RNA messaggeri
Gli RNA messaggeri, o mRNA, sono le macromolecole che contengono l’informazione per la sintesi delle proteine.
Silencer (silenziatore)
Regione nel genoma in grado di spegnere l’espressione di un gene.
Enhancer (intensificatore)
Regione nel genoma in grado di potenziare l’espressione di un gene.
Promotore
Regione nel genoma da cui parte la trascrizione (espressione) di un gene.
Organulo
Compartimento all’interno della cellula, dotato di membrana, con fuzioni specializzate (per esempio, il mitocondrio, che è l’organulo deputato alla respirazione cellulare e alla produzione di energia)
Metadato
In bioinformatica, un metadato è un gruppo di informazioni che descrive una serie di dati (per esempio dove, come, quando un campione relativo ad uno specifico dato è stato collezionato, quali erano le condizioni ambientali ecc).
Bibliografia
- Sanger F, Nicklen S, Coulson AR. DNA sequencing with chain-terminating inhibitors. Proc. Natl. Acad. Sci. U. S. A. 1977; 74:5463–5467.
- Maxam AM, Gilbert W. A new method for sequencing DNA. Proc. Natl. Acad. Sci. 1977; 74:560–564.
- Weiss MC, Preiner M, Xavier JC, et al. The last universal common ancestor between ancient Earth chemistry and the onset of genetics. PLOS Genet. 2018; 14:e1007518.
- Alföldi J, Lindblad-Toh K. Comparative genomics as a tool to understand evolution and disease. Genome Res. 2013; 23:1063–1068.
- Koboldt DC, Steinberg KM, Larson DE, et al. The next-generation sequencing revolution and its impact on genomics. Cell 2013; 155:27–38.
- Citterich MH, Ferrè F, Pavesi G, Pesole P, Romualdo C. Fondamenti di bioinformatica, Zanichelli, 2018.
- The Cost of Sequencing a Human Genome. Genome.gov.
- Sanger F, Coulson AR, Friedmann T, et al. The nucleotide sequence of bacteriophage φX174. J. Mol. Biol. 1978; 125:225–246.
- Kanz C, Aldebert P, Althorpe N, et al. The EMBL Nucleotide Sequence Database. Nucleic Acids Res. 2005; 33:D29–D33.
- Benson DA, Karsch-Mizrachi I, Lipman DJ, et al. GenBank. Nucleic Acids Res. 2007; 35:D21–D25.
- Lipman D, Pearson W. Rapid and sensitive protein similarity searches. Science 1985; 227:1435–1441.
- Altschul SF, Gish W, Miller W, et al. Basic local alignment search tool. J. Mol. Biol. 1990; 215:403–410.
- Applications of next-generation sequencing.
- Simpson JT, Pop M. The Theory and Practice of Genome Sequence Assembly. Annu. Rev. Genomics Hum. Genet. 2015; 16:153–172.
- Yandell M, Ence D. A beginner’s guide to eukaryotic genome annotation. Nat. Rev. Genet. 2012; 13:329–342.
- Leinonen R, Akhtar R, Birney E, et al. The European Nucleotide Archive. Nucleic Acids Res. 2011; 39:D28–D31.
- Karolchik D, Hinrichs AS, Kent WJ. The UCSC Genome Browser. Curr. Protoc. Hum. Genet. Editor. Board Jonathan Haines Al 2011; CHAPTER:Unit18.6.
- Ensembl 2018.
- Dudas G, Carvalho LM, Bedford T, et al. Virus genomes reveal factors that spread and sustained the Ebola epidemic. Nature 2017; 544:309–315.
- Faria NR, Azevedo R do S da S, Kraemer MUG, et al. Zika virus in the Americas: Early epidemiological and genetic findings. Science 2016; 352:345–349.
- Gostin LO. Our Shared Vulnerability to Dangerous Pathogens. Med. Law Rev. 2017; 25:185–199
- Braden CR, Dowell SF, Jernigan DB, et al. Progress in global surveillance and response capacity 10 years after severe acute respiratory syndrome. Emerg. Infect. Dis. 2013; 19:864–869
- Nichol ST, Spiropoulou CF, Morzunov S, et al. Genetic identification of a hantavirus associated with an outbreak of acute respiratory illness. Science 1993; 262:914–917
- Holmes EC, Zhang LQ, Robertson P, et al. The molecular epidemiology of human immunodeficiency virus type 1 in Edinburgh. J. Infect. Dis. 1995; 171:45–53
- Popovich KJ, Snitkin ES. Whole Genome Sequencing-Implications for Infection Prevention and Outbreak Investigations. Curr. Infect. Dis. Rep. 2017; 19:15
- Kamelian K, Montoya V, Olmstead A, et al. Phylogenetic surveillance of travel-related Zika virus infections through whole-genome sequencing methods. Sci. Rep. 2019; 9:16433
- Kugelman JR, Wiley MR, Mate S, et al. Monitoring of Ebola Virus Makona Evolution through Establishment of Advanced Genomic Capability in Liberia. Emerg. Infect. Dis. 2015; 21:1135–1143
- Guthrie JL, Gardy JL. A brief primer on genomic epidemiology: lessons learned from Mycobacterium tuberculosis. Ann. N. Y. Acad. Sci. 2017; 1388:59–77
- Grenfell BT, Pybus OG, Gog JR, et al. Unifying the epidemiological and evolutionary dynamics of pathogens. Science 2004; 303:327–332
- Drummond AJ, Suchard MA, Xie D, et al. Bayesian phylogenetics with BEAUti and the BEAST 1.7. Mol. Biol. Evol. 2012; 29:1969–1973
- Wu F, Zhao S, Yu B, et al. A new coronavirus associated with human respiratory disease in China. Nature 2020; 579:265–269
- Carter LJ, Garner LV, Smoot JW, et al. Assay Techniques and Test Development for COVID-19 Diagnosis. ACS Cent. Sci. 2020; 6:591–605
- CDC. Coronavirus Disease 2019 (COVID-19). Cent. Dis. Control Prev. 2020;
- Data – COG-UK Consortium.
- SeqCOVID – Genomic epidemiology of SARS-CoV-2 in Spain.
- Gudbjartsson DF, Helgason A, Jonsson H, et al. Early Spread of SARS-Cov-2 in the Icelandic Population. medRxiv 2020; 2020.03.26.20044446
- Walker A, Houwaart T, Wienemann T, et al. Genetic structure of SARS-CoV-2 reflects clonal superspreading and multiple independent introduction events, North-Rhine Westphalia, Germany, February and March 2020. Euro Surveill. Bull. Eur. Sur Mal. Transm. Eur. Commun. Dis. Bull. 2020; 25:
- Genomics of Indian SARS-CoV-2: Implications in genetic diversity, possible origin and spread of virus | medRxiv.
- Shu Y, McCauley J. GISAID: Global initiative on sharing all influenza data – from vision to reality. Eurosurveillance 2017; 22:30494https://dx.doi.org/10.2807%2F1560-7917.ES.2017.22.13.30494.
- Butler D. Flu researchers slam US agency for hoarding data. Nature 2005; 437:458–459.
- Butler D. Swine flu goes global. Nature 2009; 458:1082–1083.
- The fight against bird flu. Nature 2013; 496:397.
- Research Data Alliance. Final release: COVID-19 guidelines. 2020.
- Chiara M, Horner DS, Gissi C, et al. Comparative genomics provides an operational classification system and reveals early emergence and biased spatio-temporal distribution of SARS-CoV-2. bioRxiv 2020; 2020.06.26.172924.
- Rambaut A, Holmes EC, Hill V, et al. A dynamic nomenclature proposal for SARS-CoV-2 to assist genomic epidemiology. bioRxiv 2020; 2020.04.17.046086.
- Galaxy and HyPhy developments teams, Nekrutenko A, Kosakovsky Pond SL. No more business as usual: agile and effective responses to emerging pathogen threats require open data and open analytics. 2020.
- Hadfield J, Megill C, Bell SM, et al. Nextstrain: real-time tracking of pathogen evolution. Bioinforma. Oxf. Engl. 2018; 34:4121–4123.
- Argimón S, Abudahab K, Goater RJE, et al. Microreact: visualizing and sharing data for genomic epidemiology and phylogeography. Microb. Genomics 2016.
- Korber B, Fischer WM, Gnanakaran S, et al. Tracking Changes in SARS-CoV-2 Spike: Evidence that D614G Increases Infectivity of the COVID-19 Virus. Cell 2020.
- Commission on a Global Health Risk Framework for the Future, National Academy of Medicine, Secretariat. The Neglected Dimension of Global Security: A Framework to Counter Infectious Disease Crises. 2016.
- Mandl KD, Overhage JM, Wagner MM, et al. Implementing Syndromic Surveillance: A Practical Guide Informed by the Early Experience. J. Am. Med. Inform. Assoc. JAMIA 2004; 11:141–150
- Aslam AA, Tsou M-H, Spitzberg BH, et al. The reliability of tweets as a supplementary method of seasonal influenza surveillance. J. Med. Internet Res. 2014; 16:e250.
- Hulth A, Rydevik G, Linde A. Web queries as a source for syndromic surveillance. PloS One 2009; 4:e4378.
- Brownstein JS, Freifeld CC, Madoff LC. Digital disease detection–harnessing the Web for public health surveillance. N. Engl. J. Med. 2009; 360:2153–2155, 2157.
- Zinszer K, Morrison K, Verma A, et al. Spatial Determinants of Ebola Virus Disease Risk for the West African Epidemic. PLOS Curr. Outbreaks 2017.
- Majumder MS, Santillana M, Mekaru SR, et al. Utilizing Nontraditional Data Sources for Near Real-Time Estimation of Transmission Dynamics During the 2015-2016 Colombian Zika Virus Disease Outbreak. JMIR Public Health Surveill. 2016.
- Khoury MJ, Iademarco MF, Riley WT. Precision Public Health for the Era of Precision Medicine. Am. J. Prev. Med. 2016; 50:398–401.
- Kolbert E. Pandemics and the Shape of Human History. New Yorker.
- Yozwiak NL, Schaffner SF, Sabeti PC. Data sharing: Make outbreak research open access. Nature 2015; 518:477–479.
- Tam V, Patel N, Turcotte M, et al. Benefits and limitations of genome-wide association studies. Nat. Rev. Genet. 2019; 20:467–484.
- The COVID-19 Host Genetics Initiative. The COVID-19 Host Genetics Initiative, a global initiative to elucidate the role of host genetic factors in susceptibility and severity of the SARS-CoV-2 virus pandemic. Eur. J. Hum. Genet. 2020; 28:715–718.
- Ellinghaus D, Degenhardt F, Bujanda L, et al. The ABO blood group locus and a chromosome 3 gene cluster associate with SARS-CoV-2 respiratory failure in an Italian-Spanish genome-wide association analysis. medRxiv 2020; 2020.05.31.20114991.
- Ellinghaus D, Degenhardt F, Bujanda L, et al. Genomewide Association Study of Severe Covid-19 with Respiratory Failure. N. Engl. J. Med. 2020.
- Al-Zaman MS. COVID-19-related Fake News in Social Media. medRxiv 2020; 2020.07.06.20147066.
- [agg. 17/11/2020] Plante, J. A. et al. Spike mutation D614G alters SARS-CoV-2 fitness. Nature 1–9 (2020) [fine agg.]



 Scarica il PDF dell'articolo
Scarica il PDF dell'articolo
Commenti