di Barbara Illi1, Matteo Chiara2,3 e Graziano Pesole3,4
1 Istituto di Biologia e Patologia Molecolari, Consiglio Nazionale delle Ricerche (IBPM-CNR), c/o Dipartimento di Biologia e Biotecnologie “Charles Darwin”, Sapienza Università di Roma.
2 Dipartimento di Bioscienze, Università degli Studi di Milano
3 Istituto di Biomembrane Bioenergetica e Biotecnologie Molecolari, Consiglio Nazionale delle Ricerche (IBIOM-CNR)
4 Dipartimento di Bioscienze, Biotecnologie e Biofarmaceutica, Università degli Studi di Bari A. Moro
Indice dei contenuti
- Introduzione
- Tessuti bersaglio
- Trasmissione del CoV-2
- La diagnosi della COVID-19
- Diagnosi con strumenti molecolari
- Uso dell’intelligenza artificiale
- Conclusioni
“Per i non esperti”
Bibliografia
Introduzione
Dall’inizio della pandemia, la nostra visione della COVID-19 e dello stesso SARS-CoV-2 (CoV-2) è cambiata in maniera costante. Le caratteristiche cliniche della COVID-19 vanno da nessun sintomo, a sintomi lievi, fino al collasso organico, dovuto a una risposta immunitaria eccessiva (Figura 1).
Figura 1 Panoramica dei sintomi di COVID-19.
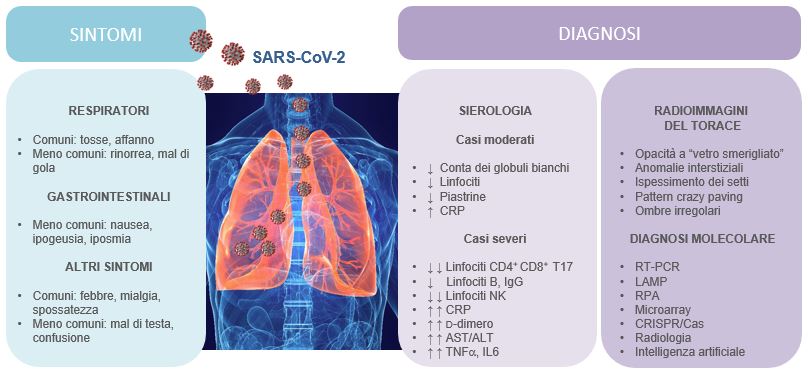
Abbreviazioni: PCR, proteina C reattiva; AST, aspartato amminotrasferasi; ALT, alanina amminotrasferasi; TNF-,fattore di necrosi del tumore; IL6, interleuchina 6; RT-PCR, Reverse Transcription-Polymerase Chain Reaction; LAMP, Loop-mediated isothermal AMPlification; RPA, Recombinase Polymerase Amplification; CT, Chest Tomography (tomografia del torace); IA, intelligenza artificiale. [Basato su: Tu et, al Int J Mol Sci, 2020; Illustrazione in centro: SciePro/Shutterstock]
Questa varietà di manifestazioni cliniche dipende, probabilmente, dalla carica virale a cui il paziente è stato esposto, da patologie pre- e co-esistenti, ma anche da fattori come età, sesso e genetica del sistema immunitario (in particolare del sistema HLA). In effetti, è stato dimostrato che nei casi gravi, la carica virale media è 60 volte superiore a quella dei casi lievi1. Il CoV-2 può infettare molti tessuti e può essere isolato da diversi fluidi biologici, che rappresentano il materiale di partenza per la diagnosi di COVID-19.
Tessuti bersaglio
I tessuti che esprimono l’ACE2 sono i bersagli diretti dell’infezione da CoV-2. Questi includono non solo le cellule epiteliali delle vie respiratorie superiori e inferiori, ma anche le cellule vascolari (come i periciti)2 e le cellule epiteliali di bocca, gengiva, lingua e della mucosa orale3. L’espressione di ACE2 è stata rilevata anche nelle ghiandole salivari4. È stato dimostrato che cellule epiteliali del dotto minore delle ghiandole salivari del macaco rhesus possono essere infettate dal SARS-CoV5, perciò è ipotizzabile che anche il SARS-CoV-2 si comporti nello stesso modo. Inoltre, l’ACE2 è sintetizzato anche dalle cellule dell’esofago e da altre cellule del tratto gastrointestinale, inclusi gli enterociti. In effetti, è stato osservato di recente che questi ultimi esprimono alti livelli di ACE2 e possono essere infettati da SARS-CoV-2 (v.Figura 3A di Lamars et al. Science 2020, in cui mediante immunofluorescenza bianca è evidenziata la presenza della proteina del nucleocapside virale, NP, negli enterocit; in verde, colorata con la falloidina, l’actina che compone il citoscheletro)6.
Trasmissione del CoV-2
Come ogni virus respiratorio, la trasmissione di SARS-CoV-2 avviene per emissione di particelle dette droplet, principalmente starnutendo o tossendo (Figura 2). Laddove una persona emette circa 75 000 particelle per ogni colpo di tosse7, l’ordinario parlare può produrre un numero di droplet nel complesso ancora più alto, rappresentando, pertanto un veicolo di infezione. Inoltre, è stato mostrato che questo numero differisce a seconda dell’intensità del volume della voce.
Figura 2 Relazione tra dimensione dei droplets e loro dispersione nell’aria.
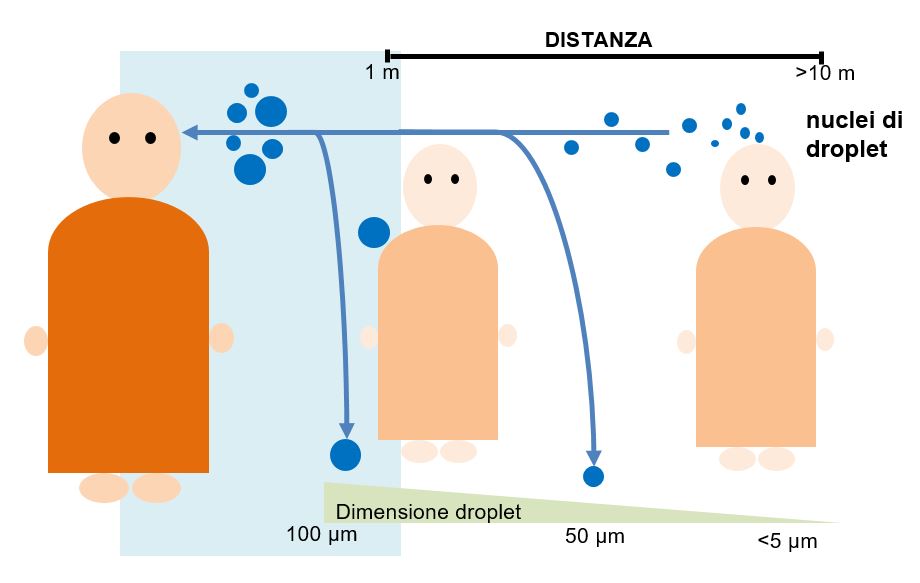
[Basato su International Federation of Infection control, www.theific.org, 2016].
La dimensione delle droplet può variare da 1 a 500 μm, ma, dal momento che le droplet piccole evaporano velocemente e quelle grandi sono soggette a velocità di caduta gravitazionale, le droplet di diametro medio (da 30 a 50 μm) sono quelle che raggiungono la distanza massima orizzontale, stimata in meno di 1 m durante il respiro e fino a 6 m per uno starnuto8.
Parlare ad alta voce può produrre 2600 particelle al secondo. Queste particelle si disidratano velocemente (producendo i cosidetti nuclei droplet). Per esempio, le particelle di 12-21 μm, con un volume da 60 a 320 nL, si disidratano rapidamente fino a raggiungere un diametro di 4 μm. La vita media di queste particelle è 8 minuti e, considerando che la carica virale del CoV-2 nel fluido orale di circa 7 · 106 copie per millilitro (mL; la carica virale media è stata stimata in 2,35 · 1011 copie/mL9), è stato calcolato che 1 minuto di conversazione ad alta voce può generare 1000 droplet contenti virioni, che permangono in aria dagli 8 ai 14 minuti. La probabilità che le particelle piccole contengano virioni cala dal 37% per droplet di 50 μm di diametro allo 0,37% per particelle di 10 μm, mentre la probabilità per queste ultime di contenere più di un virione è irrilevante10.
Le particelle più piccole possono rimanere indefinitamente nell’aria; tuttavia la probabilità per particelle disidratate di 1 μm (partendo da droplet di 3 μm) di contenere un virione è dello 0,01%10. Sebbene il numero di particelle emesse aumenti con l’aumentare del volume della voce, la distribuzione delle dimensioni delle particelle non cambia (v. Figura 2B in Asadi et al., Sci Rep 2020, il grafico mostra la distribuzione delle droplet per dimensione durante l’emissione di una lettera, per otto volumi di voce diversi, ripetuta per sei volte; abbreviazioni usate: Arms, root mean square Amplitude, ossia scarto quadratico medio dell’estensione vocale; F0, frequenza fondamentale, ovvero la frequenza alla quale le corde vocali vibrano mentre si parla).
Inoltre, è stata dimostrata l’esistenza di super-emettitori di droplet, che potrebbero spiegare la presenza di super-diffusori di SARS-CoV-211. Si è anche ipotizzata una trasmissione fecale, un argomento ancora dibattuto. Sono state trovate copie dell’RNA virale nelle feci, indipendentemente da tamponi nasofaringei negativi o positivi e dall’assenza/presenza di sintomi12. La rilevazione dell’RNA virale nelle feci non significa necessariamente che queste contengano virioni infettivi. Tuttavia, la presenza di virioni vitali nelle feci è stata riportata in seguito a osservazioni con il microscopio elettronico13. La carica virale nei campioni di feci è stata determinata in 0,7–0,6 copie di RNA virale per millilitro, rispettivamente nei pazienti con e senza diarrea14.
Questo è un punto importante, poiché il CoV-2 potrebbe infettare anche in presenza di carica virale bassa. Per cui, sebbene si pensi che la carica virale nelle feci dovrebbe essere bassa, la possibilità di una trasmissione fecale non deve essere sottovalutata. Inoltre, l’RNA virale è stato trovato anche nel sangue e nelle urine13 e la contaminazione è noto che possa avvenire anche per contatto con superfici contaminate. A tal proposito, l’emivita delle particelle è diversa a seconda del materiale15.
La diagnosi della COVID-19
La Figura 1 mostra una varietà di metodi oggi impiegati per la rilevazione del CoV-2 e che consentono la diagnosi della COVID-19.
Diagnosi con strumenti molecolari
Metodi basati sulla reazione a catena della polimerasi (PCR, Polymerase Chain Reaction)
Inizialmente, l’identificazione molecolare del CoV-2 si è basata sul processo di trascrizione inversa associato alla PCR (RT-PCR, Reverse Transcription-Polymerase Chain Reaction) da tamponi nasofaringei od orofaringei, che può essere eseguita usando il metodo SYBRGreen® o TaqMan® (Video 1).
All’inizio della pandemia, la Food and Drug Administration (FDA) ha approvato un test commerciale per la rilevazione del CoV-2 della ditta Roche (cobas® SARS-CoV-2). Questo test richiede 3,5 ore; più avanti è stato approvato un altro test, più veloce, l’Xpert® Xpress SARS-CoV-2 della ditta Cepheid Inc (US), con cui si ottiene il risultato in 45 minuti.
Video 1 Come funziona il sistema TaqMan (video in inglese).
Amplificazione isotermica mediata da loop (LAMP)
L’amplificazine mediata da loop (LAMP, Loop-mediated isothermal AMPlification) è più economica e veloce (Video 2). Inoltre, usando inneschi multipli, è più sensibile e specifica. La rilevazione finale dei prodotti può essere fatta o per elettroforesi su gel di agarosio e/o per real time PCR (RT-LAMP). L’RT-LAMP è già stata usata per rilevare la presenza di altri coronavirus (SARS-CoV16, MERS-CoV17, HCoV-NL6318); ha permesso di identificar fino a 3,4 copie del MERS-CoV17 e 0,01 unità formanti placche (PFU, Plaque Forming Unit) di SARS-CoV16. Per il SARS-CoV-2, il limite di rilevazione è stato stimato in 2 · 102 copie per reazione19. Recentemente, questa tecnica è stata modificata facendo seguire il sequenziamento all’amplificazione isotermica (Video 3).
Video 2 Amplificazione mediata da loop (video in inglese).
Video 3 Sequenziamento mediante sintesi (video in inglese).
In particolare, i prodotti della reazione dell’RT-LAMP vengono frammentati dalla trasposasi Tn5 che contestualmente aggiunge adattatori che contengono specifici “codici” (gli indici) ai frammenti20. Si possono usare fino a 96 indici, consentendo l’analisi di 96 campioni in parallelo (Figura 3). Successivamente, i frammenti vengono amplificati dalla DNA polimerasi e sequenziati. Questa metodica si è dimostrata una valida alternativa alla versione precedente di RT-LAMP20.
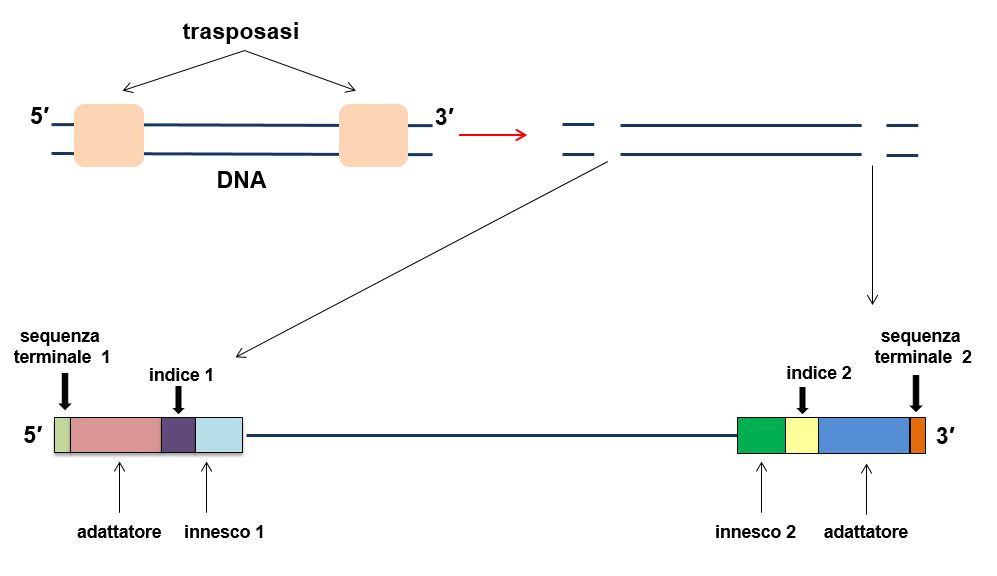
Amplificazione con polimerasi e ricombinasi (RPA)
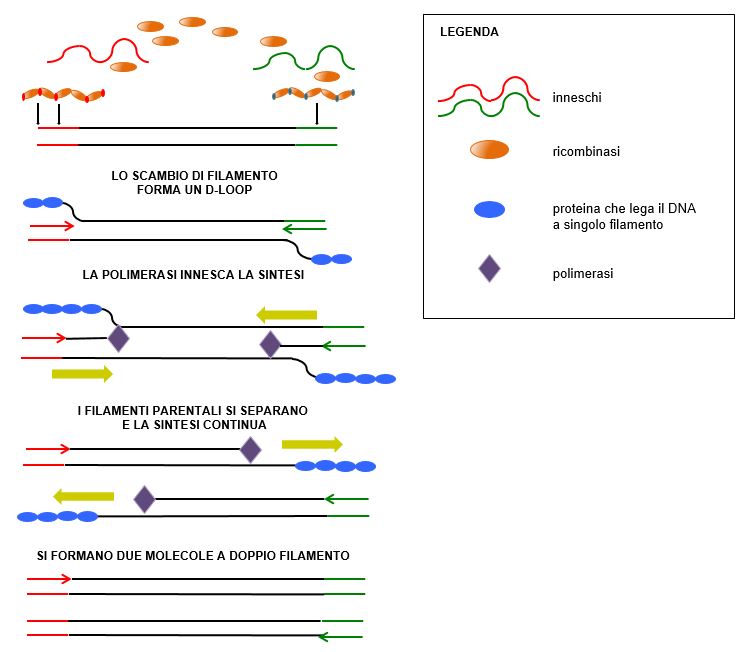
L’amplificazione con ricombinasi e polimerasi (RPA, Recombinase Polymerase Amplification) è un’evoluzione dei metodi basati sulla PCR. Per questa tecnica si usano due inneschi opposti, come per la PCR, complementari alla sequenza che si vuole rilevare e complessati con l’enzima ricombinasi: in questo modo si forma un D-loop nella sequenza bersaglio a doppio filamento. Gli inneschi sono, poi, estesi da una DNA polimerasi mesofilica (ossia, stabile a temperature moderate; Figura 4). Se si aggiungono sonde fluorescenti, la reazione di RPA può essere monitorata in tempo reale. Questo metodo è stato applicato di recente per rilevare la presenza del CoV-2 in 20 minuti, con i primi risultati visibili dopo 7 minuti21.
Figura 4 Schema di una reazione di RPA.
Microarray
Anche i microarray possono essere usati per rilevare il CoV-2. Anche altri coronavirus sono stati rilevati con questo metodo22. In pratica, l’RNA virale viene retrotrascritto in cDNA e categorizzato mediante sonde specifiche. I cDNA marcati sono ibridati con sonde specifiche fissate su un microarray. Una serie di lavaggi eliminano, poi, i cDNA che non ibridati.
Metodi basati sul sequenziamento di nuova generazione (NGS)
I metodi basati sul sequenziamento di nuova generazione (NGS, Next Generation Sequencing) possono rilevare simultaneamente più genomi virali, grazie alla possibilità di sequenziare tutto l’insieme degli RNA di una cellula infetta. L’analisi bioinformatica delle sequenze ottenute, partendo dall’allineamento delle sequenze con quelle disponibili in GenBank, porta alla categorizzazione di nuovi virus. Questi metodi sono anche (e soprattutto) usati per identificare varianti genomiche e per studi di filogenesi (v. Spillover e pandemia di COVID-19: una prospettiva genomica). Per esempio, di recente, l’analisi di 7666 genomi virali di CoV-2 in diversi Paesi ha permesso l’identificazione di mutazioni ricorrenti, avvenute indipendentemente nel tempo (omoplasie), in specifiche regioni genomiche del CoV-223.
Metodi basati sulla tecnologia CRISPR/Cas
L’avanzamento tecnologico più recente per la diagnosi molecolare di CoV-2 è rappresentato dalla piattaforma CARMEN (Combinatorial Arrayed Reactions for Multiplexed Evaluation of Nucleic acid), che si basa sulla tecnologia CRISPR/Cas24. Il sistema CRISPR/Cas è basato sull’uso di un RNA guida complessato con la Cas. Se l’RNA guida lega una sequenza complementare in un acido nucleico bersaglio, l’enzima Cas si attiva e taglia il bersaglio.
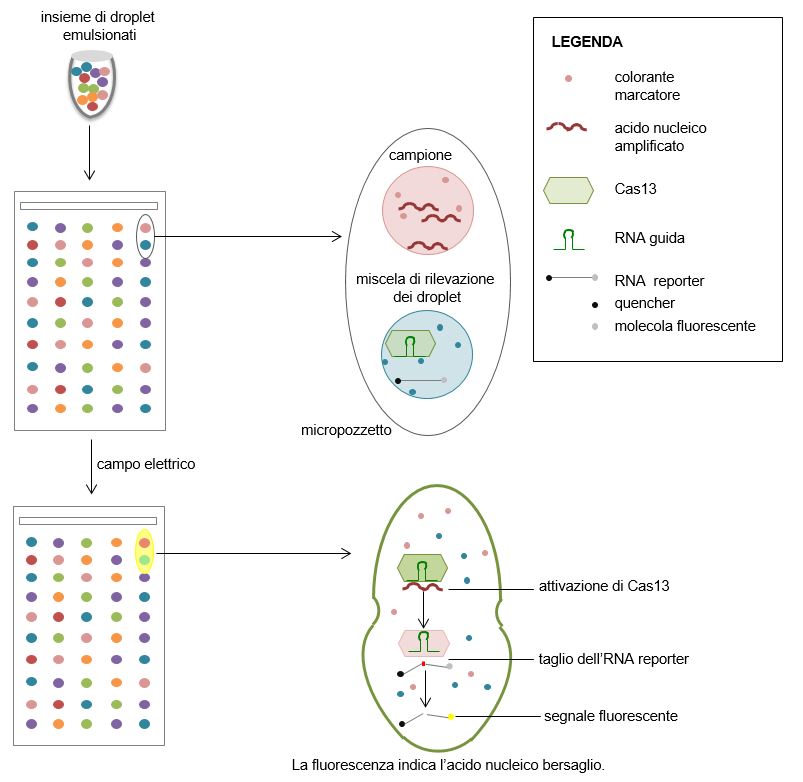
La piattaforma CARMEN sfrutta Cas13, che taglia solo l’RNA e non il DNA. In questo metodo, la Cas13 taglia un RNA reporter fluorescente in maniera non specifica quando è attivato dal riconoscimento di una sequenza complementare. La PCR o l’RPA sono necessari nella prima fase, per amplificare l’RNA virale (se presente). Un colorante fluorescente è mescolato al campione per conferire all’RNA un codice colore specifico. Sono generate per tutte le diveres reazioni di amplificazione delle gocce (droplet) da 1 nanolitro, emulsionate con olio. È anche prodotta un’altra serie di droplet con colori unici. Questi droplet colorati contengono un RNA reporter fluorescente e la Cas13 legata a un RNA guida, necessario per rilevare l’RNA virale bersaglio. I droplet mescolati (in un’unica provetta) sono caricati su un chip contenente dei micropozzetti, capaci di alloggiare soltanto due droplet. Sul chip, ogni acido nucleico bersaglio è esposto probabilisticamente a ciascuna miscela di rilevazione, in replicati multipli, in diverse posizioni. L’esposizione a un campo elettrico, mescolando la coppia di droplet, innesca la reazione di rilevazione. Se la Cas13, complessata con l’RNA guida, riconosce una sequenza virale amplificata nello stesso pozzetto, si attiva e genera una fluorescenza dall’RNA reporter, a causa dell’attività di taglio non specifica, che viene rilevata (Figura 5). Questa piattaforma è estremamente innovativa e permette di rilevare più di un virus per ogni esperimento. È stato dimostrato che è in grado di distinguere il CoV-2 da altri coronavirus umani, quali SARS-CoV and MERS-CoV24.
Figura 5 Rappresentazione grafica della piattaforma CARMEN.
Uso dell’intelligenza artificiale
La rilevazione mediante RT-PCR è, a oggi, il metodo di riferimento diagnosticare un’infezione da CoV-2, ma può essere un processo lungo, in quanto può richiedere test multipli, per escludere i falsi negativi. Le tempistiche necessarie per questo tipo di diagnosi non sempre si conciliano con le esigenze di una situazione di emergenza. L’applicazione di approcci diagnostici complementari o alternativi può, perciò, costituire uno molto strumento utile per il triage prima dell’ottenimento della diagnosi definitiva.
La diagnosi rapida dell’infezione da Cov-2 nelle strutture ospedaliere o in altri contesti critici per il contenimento della pandemia (per esempio, porti e aeroporti) è fondamentale per minimizzare il rischio di trasmissione virale ad altri soggetti e anche perché un esito falso negativo potrebbe ritardare il trattamento di un soggetto sintomatico che potrebbe vedere peggiorare velocemente il suo quadro clinico.
La tomografia computerizzata (TC) non può essere applicata per diagnosticare i pazienti con una forma lieve della malattia, che non sempre manifestano i tipici segni radiologici (v. sopra). Inoltre, non tutti gli istituti ospedalierei potrebbero avere a disposizione dei radiologi con esperienza nell’imaging toracico. Infine come tutte le attività umane, la diagnosi basata sulla TC è un processo con un certo tasso di errore, soggetto all’interpretazione, non sempre riproducibile, e quindi non è completamente affidabile. Proprio per questo motivo negli ultimi anni vari autori hanno suggerito l’uso di strumenti di intelligenza artificiale, allo scopo di migliorare i processi diagnostici basati sulle analisi delle immagini.
L’intelligenza artificiale (IA o AI, Artificial Intelligence) e le tecnologie correlate trovano sempre maggiore applicazione nella nostra società, e hanno certamente il potenziale per migliorare anche alcuni aspetti legati alla diagnosi dei pazienti. Con il termine AI non si intende una singola tecnologia, ma piuttosto un insieme di tecniche computazionali. Le metodiche di questo tipo maggiormente usate per la clinica e la diagnostica sono le cosiddette tecniche di machine learning o apprendimento automatico.
L’apprendimento automatico è una tecnica computazionale in grado di generare modelli per la soluzione di problemi che sono in grado di apprendere direttamente dai dati. L’apprendimento automatico è una tecnica ampia, alla base di molti approcci all’intelligenza artificiale. Esistono numerose implementazioni diverse dei metodi di apprendimento automatico. L’applicazione più comune in ambito clinico è l’implementazione di metodi per la medicina di precisione, una disciplina che richiede la capacità di prevedere quali protocolli di trattamento hanno maggiore probabilità di avere successo su determinati pazienti in base ai vari attributi dei pazienti e al contesto del trattamento.
La grande maggioranza delle applicazioni di questo tipo richiede, per addestrare un modello di apprendimento, un insieme di dati già classificati (per esempio, caratteristiche dei pazienti a seguito di insorgenza della malattia) da cui il metodo computazionale è in grado di derivare le caratteristiche salienti; questo approccio è chiamato apprendimento supervisionato in quanto l’algoritmo impara dagli esempi noti.
Uno degli algoritmi più di successo applicato ai problemi di apprendimento automatico è la rete neurale: una tecnologia disponibile dagli anni ’60 del Novecento che è stata utilizzata nella ricerca sanitaria per diversi decenni, per esempio per classificare i pazienti o predire possibili fattori di rischio per una malattia. Nella sua formulazione più semplice, la rete neurale visualizza i problemi in termini di relazioni tra l’input e l’output. Queste relazioni vengono descritte da determinate caratteristiche, cui la rete associa un peso ottimale. Più una caratteristica dell’input è fondamentale per descrivere/ottenere l’output, maggiore è il peso a essa assegnato. Per esempio, in un caso di classificazione di pazienti, l’input potrebbero essere i soggetti dell’indagine (per esempio, pazienti e controlli sani), da cui il sistema determina automaticamente la diagnosi (per esempio, malato o sano) sulla base delle loro caratteristiche (per esempio, analisi ematiche o altri parametri clinici).
Le forme più avanzate di machine learning sono oggi note come deep learning: modelli di rete neurale con un numero elevatissimo di strati, che analizzano diversi sottoinsiemi di problemi e che mostrano una topologia molto articolata. Un sistema di deep learning potrebbe contenere e integrare migliaia di reti neurali che si occupano di risolvere diversi problemi specifici, per poi integrare i risultati in una singola predizione. Questi approcci vengono sempre più applicati nella diagnostica medica, poiché sono più efficienti dell’occhio umano nel valutare in maniera esaustiva le complesse immagini radiologiche. L’uso di questi approcci sembra promettere una maggiore precisione nella diagnosi rispetto alla precedente generazione di strumenti e metodi.
Alcune applicazioni basate su metodi di intelligenza artificiale sono state recentemente usate anche per la diagnosi della COVID-19. Un gruppo di ricercatori dell’Icahn School of Medicine di Manhattan è stato in grado di sviluppare un algoritmo di apprendimento automatico, basato su un una rete neurale, che applica principi di intelligenza artificiale a sostegno delle metodiche molecolari. Questo algoritmo è stato progettato per favorire/velocizzare la diagnosi in tempi rapidi di pazienti che si sospetta possano avere la COVID-19. Il software, oltre ad un elaborato modello per l’analisi dei dati i radiologici dei pazienti, integra nel processo di decisione la loro storia clinica, i risultati di tutti i test di laboratorio eseguiti, i diversi parametri fisiologici, i sintomi e, quando possibile, o dati riguardo a episodi di esposizione ad altri soggetti affetti, o informazioni sulle catene di contagio locali25. Gli autori dello studio hanno dimostrato che la rete neurale è più sensibile e specifica (84,3% e 82,8%) dei modelli basati solo solo sulle immagini radiologiche (83,6% e 75,9%) o sui dati clinici (80,6% e 68,3%)25. Tenendo conto di questi dati sperimentali, l’algoritmo potrebbe essere usato per identificare rapidamente le persone con il CoV-2 prima dei risultati dei test molecolari.
Conclusioni
Dal momento che la pandemia di COVID-19 non è ancora sotto controllo e sta ancora crescendo rapidamente nel mondo, è imperativo ottenere diagnosi rapide, per isolare i pazienti e relativi contatti. Una moltitudine di metodi sono stati applicati, dimostrando come differenti competenze in ambito clinico, biomolecolare e matematico-statistico possono convergere su un terreno comune. Dai metodi di RT-PCR, alle ultime frontiere della tecnologia CRISPR/Cas e dell’intelligenza artificiale, si sta facendo un enorme sforzo per assicurare non solo l’identificazione veloce e sicura degli individui con il CoV-2, ma soprattutto si può migliorare la loro prognosi e si possono definire protocolli terapeutici sempre più appropriati.
Per i non esperti
RT-PCR
L’RT-PCR è un metodo basato sulla produzione e rilevazione di copie multiple di un gene di interessepartendo dall’RNA come stampo; nel caso discusso nel testo il gene amplificato è un gene specifico del CoV-2 come N, che codifica la proteina del nucleocapside.
L’enzima trascrittasi inversa, usando come stampo l’RNA totale estratto dalla cellula, piccole sequenze (oligonucleotidi) come inneschi (primer) e nucleotidi come “mattoni”, sintetizza un singolo filamento di DNA complementare (cDNA) alle sequenze di RNA. Dopodiché, delle DNA polimerasi batteriche, resistenti al calore (come la Taq polimerasi, del Thermus acquaticus o la Pfu polimerasi del Pyrococcus furiosus), producono copie multiple del gene di interesse partendo dal cDNA e da specifici oligonucleotidi che selezionano il gene bersaglio.
I prodotti dell’amplificazione possono essere visualizzati su un gel di agarosio, che separa le molecole di DNA secondo il loro peso molecolare. Questa è, tuttavia, un’ analisi qualitativa.
Per monitorare la quantità di trascritto in un determinato campione, bisogna usare la real time PCR. Questa metodica è basata sulla misura dell’emissione di fluorescenza man mano che le molecole di DNA aumentano nel campione. Si possono usare coloranti fluorescenti che legano il DNA a doppio filamento, come il SYBRGreen® (meno specifici) o sonde specifiche fluorescenti, come le sonde Taqman® (più specifiche). Differiscono perché i coloranti che legano il DNA a doppio filamento si legano a ogni molecola, aumentando la fluorescenza del campione man mano che aumenta la quantità di molecole di DNA, mentre una sonda specifica fluorescente si lega solo a sequenze di DNA a essa complementari. In questo caso, la fluorescenza è rilasciata solo alla fine della sintesi del filamento di DNA, quando la sonda è degradata. In ogni caso, la fluorescenza è rilevata da una macchina apposita e la quantità di DNA calcolata secondo il metodo descritto Livak eSchmittgen 200126.
Bibliografia
- Liu, Y. et al. Viral dynamics in mild and severe cases of COVID-19. Lancet Infect Dis 20, 656-657, doi:10.1016/S1473-3099(20)30232-2 (2020).
- Chen, L., Li, X., Chen, M., Feng, Y. & Xiong, C. The ACE2 expression in human heart indicates new potential mechanism of heart injury among patients infected with SARS-CoV-2. Cardiovasc Res 116, 1097-1100, doi:10.1093/cvr/cvaa078 (2020).
- Xu, H. et al. High expression of ACE2 receptor of 2019-nCoV on the epithelial cells of oral mucosa. Int J Oral Sci 12, 8, doi:10.1038/s41368-020-0074-x (2020).
- Song, J. et al. Systematic analysis of ACE2 and TMPRSS2 expression in salivary glands reveals underlying transmission mechanism caused by SARS-CoV-2. J Med Virol, doi:10.1002/jmv.26045 (2020).
- Liu, L. et al. Epithelial cells lining salivary gland ducts are early target cells of severe acute respiratory syndrome coronavirus infection in the upper respiratory tracts of rhesus macaques. J Virol 85, 4025-4030, doi:10.1128/JVI.02292-10 (2011).
- Lamers, M. M. et al. SARS-CoV-2 productively infects human gut enterocytes. Science, doi:10.1126/science.abc1669 (2020).
- Lindsley, W. G. et al. Quantity and size distribution of cough-generated aerosol particles produced by influenza patients during and after illness. J Occup Environ Hyg 9, 443-449, doi:10.1080/15459624.2012.684582 (2012).
- Xie, X., Li, Y., Chwang, A. T., Ho, P. L. & Seto, W. H. How far droplets can move in indoor environments–revisiting the Wells evaporation-falling curve. Indoor Air 17, 211-225, doi:10.1111/j.1600-0668.2007.00469.x (2007).
- Wolfel, R. et al. Virological assessment of hospitalized patients with COVID-2019. Nature 581, 465-469, doi:10.1038/s41586-020-2196-x (2020).
- Stadnytskyi, V., Bax, C. E., Bax, A. & Anfinrud, P. The airborne lifetime of small speech droplets and their potential importance in SARS-CoV-2 transmission. Proc Natl Acad Sci U S A 117, 11875-11877, doi:10.1073/pnas.2006874117 (2020).
- Asadi, S. et al. Aerosol emission and superemission during human speech increase with voice loudness. Sci Rep 9, 2348, doi:10.1038/s41598-019-38808-z (2019).
- Amirian, E. S. Potential fecal transmission of SARS-CoV-2: Current evidence and implications for public health. Int J Infect Dis 95, 363-370, doi:10.1016/j.ijid.2020.04.057 (2020).
- Wang, W. et al. Detection of SARS-CoV-2 in Different Types of Clinical Specimens. JAMA, doi:10.1001/jama.2020.3786 (2020).
- Cheung, K. S. et al. Gastrointestinal Manifestations of SARS-CoV-2 Infection and Virus Load in Fecal Samples From a Hong Kong Cohort: Systematic Review and Meta-analysis. Gastroenterology, doi:10.1053/j.gastro.2020.03.065 (2020).
- van Doremalen, N. et al. Aerosol and Surface Stability of SARS-CoV-2 as Compared with SARS-CoV-1. N Engl J Med 382, 1564-1567, doi:10.1056/NEJMc2004973 (2020).
- Kim, J. H. et al. A Simple and Multiplex Loop-Mediated Isothermal Amplification (LAMP) Assay for Rapid Detection of SARS-CoV. Biochip J 13, 341-351, doi:10.1007/s13206-019-3404-3 (2019).
- Shirato, K. et al. Development of fluorescent reverse transcription loop-mediated isothermal amplification (RT-LAMP) using quenching probes for the detection of the Middle East respiratory syndrome coronavirus. J Virol Methods 258, 41-48, doi:10.1016/j.jviromet.2018.05.006 (2018).
- Pyrc, K., Milewska, A. & Potempa, J. Development of loop-mediated isothermal amplification assay for detection of human coronavirus-NL63. J Virol Methods 175, 133-136, doi:10.1016/j.jviromet.2011.04.024 (2011).
- Baek, Y. H. et al. Development of a reverse transcription-loop-mediated isothermal amplification as a rapid early-detection method for novel SARS-CoV-2. Emerg Microbes Infect 9, 998-1007, doi:10.1080/22221751.2020.1756698 (2020).
- Dao Thi, V. L. et al. A colorimetric RT-LAMP assay and LAMP-sequencing for detecting SARS-CoV-2 RNA in clinical samples. Sci Transl Med 12, doi:10.1126/scitranslmed.abc7075 (2020).
- Behrmann, O. et al. Rapid detection of SARS-CoV-2 by low volume real-time single tube reverse transcription recombinase polymerase amplification using an exo probe with an internally linked quencher (exo-IQ). Clin Chem, doi:10.1093/clinchem/hvaa116 (2020).
- Chen, Q. et al. Comprehensive detection and identification of seven animal coronaviruses and human respiratory coronavirus 229E with a microarray hybridization assay. Intervirology 53, 95-104, doi:10.1159/000264199 (2010).
- van Dorp, L. et al. Emergence of genomic diversity and recurrent mutations in SARS-CoV-2. Infect Genet Evol 83, 104351, doi:10.1016/j.meegid.2020.104351 (2020).
- Ackerman, C. M. et al. Massively multiplexed nucleic acid detection with Cas13. Nature 582, 277-282, doi:10.1038/s41586-020-2279-8 (2020).
- Mei, X. et al. Artificial intelligence-enabled rapid diagnosis of patients with COVID-19. Nat Med, doi:10.1038/s41591-020-0931-3 (2020).
- Livak, K. J. & Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods 25, 402-408, doi:10.1006/meth.2001.1262 (2001).


 Scarica il PDF dell'articolo
Scarica il PDF dell'articolo
Commenti