di Barbara Illi
Istituto di Biologia e Patologia Molecolari, Consiglio Nazionale delle Ricerche (IBPM-CNR), c/o Dipartimento di Biologia e Biotecnologie “Charles Darwin”, Sapienza Università di Roma
Indice dei contenuti
- Introduzione
- Le prime fasi dopo l’ingresso del virus: la produzione delle proteine di SARS-CoV-2
- La replicazione del genoma di SARS-CoV-2
- I salti dell’RNA polimerasi dipendente dall’RNA
- Ma le cose sono più complicate di così: le modifiche dell’RNA
- Conclusioni
“Per i non esperti”
Bibliografia
Introduzione
Il sopraggiungere dell’epidemia del nuovo coronavirus (denominato prima “novel Coronavirus 2019” o 2019-nCoV, ora SARS-CoV-2, da Severe Acute Respiratory Syndrome Coronavirus 2; la malattia causata è detta COVID-19) a fine 2019 rappresenta ancora una grande sfida per la comunità scientifica. Si sta compiendo uno sforzo enorme per raccogliere il maggior numero di informazioni nel minor tempo possibile e, infatti, nel solo periodo da gennaio 2020 a giugno 2020, sono stati pubblicati oltre 10 000 articoli scientifici sull’argomento. Si sono verificate anche altre epidemie dovute a coronavirus negli ultimi 17 anni: l’epidemia di SARS, nel 2003, e la MERS (Middle East Respiratory Syndrome), nel 2012. Tuttavia, queste due epidemie sono state così efficientemente controllate che i vaccini prodotti per limitarne la diffusione non sono stati utilizzati. Infatti, il vaccino contro il SARS-CoV si è fermato alle fasi I e II dei clinical trial1,2, mentre quello contro il MERS-CoV è ancora in fase di sperimentazione. Il SARS-CoV-2 (d’ora in poi CoV-2) è per l’80% identico a suo fratello SARS-CoV, che causa la SARS.
Questi virus condividono anche molte caratteristiche biologiche, inclusi il meccanismo e le proteine che usano per entrare nella cellula ospite. Il meccanismo di replicazione e trascrizione del genoma di SARS-CoV-2 sembra comune a quello di altri coronavirus. Allora, perché questo virus ha manifestazioni epidemiologiche e cliniche così diverse dagli altri coronavirus? Per rispondere a questa domanda, dobbiamo fare un passo indietro e analizzare il meccanismo di replicazione e trascrizione dei coronavirus.
Le prime fasi dopo l’ingresso del virus: la produzione delle proteine di SARS-CoV-2
I coronavirus hanno il genoma più grande, circa 30 kilobasi, di tutti i virus a RNA.
Una volta che il coronavirus è entrato nella cellula, il suo genoma a RNA viene immediatamente tradotto dai ribosomi e da proteine specifiche della cellula ospite. Si forma una poliproteina gigante, detta pp1ab, codificata dal gene replicasi (replicase), a partire da due regioni del genoma virale che possono essere tradotte in proteine e pertanto dette “cornici di lettura aperte” (ORF, Open Reading Frames). La pp1ab è tagliata in 16 proteine più piccole non strutturali (nsp, non structural proteins)3,4. È da notare che due delle proteine prodotte precocemente dall’RNA virale (nsp3 ed nsp5) sono coinvolte ne taglio di pp1ab. Tutte le 16 nsp sono necessarie per la replicazione dell’RNA genomico (gRNA) e la produzione dei vari RNA messaggeri (mRNA) sub-genomici virali (sgRNA, i frammenti del genoma da cui vengono tradotte le proteine virali). A valle del gene replicasi, è contenuta l’informazione per la produzione delle proteine strutturali:
- spike;
- envelope (proteina del rivestimento del virione);
- M (proteina di membrana);
- N (proteina del nucleocapside, che complessa l’RNA virale).
Queste proteine “impacchettano” il genoma virale e sono necessarie alla produzione di nuovi virioni. Inoltre, sempre dal tratto a valle del gene replicasi, sono prodotte sei proteine accessorie (3a, 6, 7a, 7b, 8, 10) il cui ruolo non è ancora del tutto chiaro. Nella Figura 1 sono rappresentate in modo schematico le proteine sintetizzate a partire dall’RNA del CoV-2 e come si assemblano per formare il virione5.
Figura 1 Struttura del genoma di SARS-CoV-2
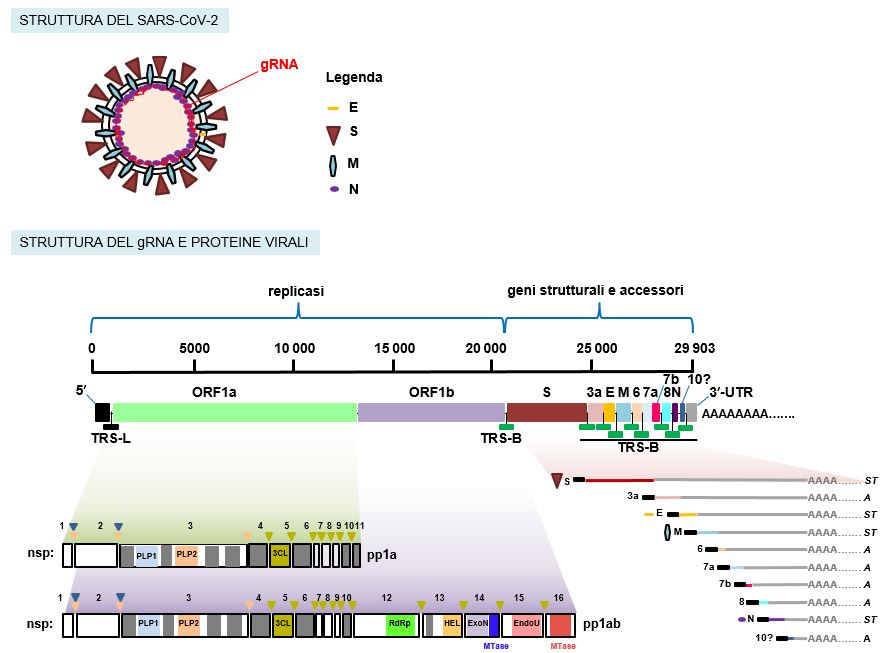
La maggior parte del genoma è occupato dal gene replicasi, che codifica per 16 nsp, prodotte dal taglio della poliproteina pp1ab. A valle, è presente l’informazione per le proteine strutturali e accessorie. Come si può osservare nella figura, oltre alla proteina pp1ab, viene prodotta anche una proteina più corta, pp1a, a partire dalla sola sequenza ORF1A. Abbreviazioni: L, Leader; TRS-L, Transcription Regulatory Sequence At The Leader (sequenza regolatoria della trascrizione al Leader); TRS-B, Transcription Regulatory Sequence At The Body (sequenza regolatoria della trascrizione all’interno del corpo del genoma); ORF, Open Reading Frame (cornice di lettura aperta); S, Spike; E, Envelope (proteina del rivestimento virale); M, Membrane (proteina della membrana); N, Nucleocapsid (proteina del nucleocapside che complessa il gRNA); le sigle 3a, 6, 7a, 7b, 8, 10 indicano i geni accessori; nsp, non structural protein; PLP, Papain-Like Protease (proteasi simile alla papaina); 3CL, Chymotrypsin-Like Protease (proteasi simile alla chimotripsina); RdRp, RNA-dependent RNA polymerase (RNA polimerasi dipendente dall’RNA); HEL, HELicase (elicasi); ExoN, ExoNuclease (esonucleasi); EndoU, EndonUclease (endonucleasi); Mtase, Methyltransferase (metiltrasferasi); UTR, UnTranslated Region (regione non tradotta); ST, proteina strutturale; A, proteina accessoria. [Basato su Kim et al., Cell, 2020 and Sola et al., Ann Rev Virol, 2015].
La replicazione del genoma di SARS-CoV-2
L’RNA dei coronavirus è, di fatto, un mRNA molto lungo. Si tratta di un filamento singolo positivo, ossia viene letto dalle strutture deputate alla traduzione nella direzione cosiddetta 5′→3′ (Figura 2A).
La replicazione dell’RNA, cioè la produzione di più copie del genoma virale, è un processo continuo. Il nuovo gRNA viene prodotto per intero grazie alla sintesi di un filamento intermedio negativo (che decorre in direzione 3′→5′; linea arancione tratteggiata nella Figura 2A), che serve da stampo per produrre un nuovo gRNA positivo. Questo processo coinvolge principalmente la proteina nsp12, che è un’RNA polimerasi dipendente dall’RNA (RdRp), e che sintetizza il primo filamento negativo legandosi all’estremità 3′ del gRNA. Il legame è possibile grazie anche alla formazione di strutture complesse che flettono l’RNA in modo da facilitarne la replicazione grazie all’avvicinamento di alcune porzioni (Figura 2B).
Figura 2 La replicazione dell’RNA di SARS-CoV-2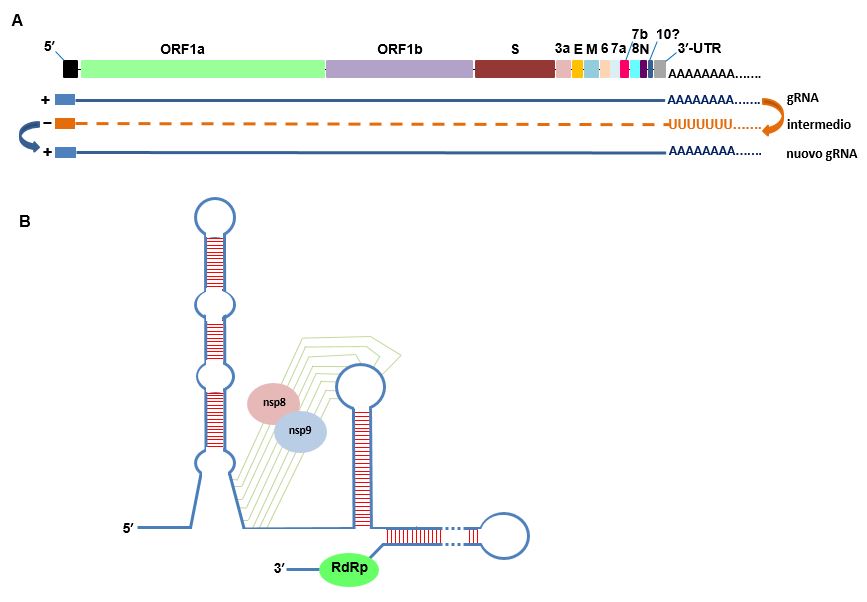
A Sintesi continua del gRNA a partire da uno stampo intermedio negativo. B Esempio di formazione di una struttura complessa dell’RNA, mediata da nsp8 ed nsp9, che consente il legame di RdRp all’estremità 3′ dell’RNA e la sintesi del filamento negativo. [Basato su Kim et al., Cell, 2020 and Sola et al, RNA Biol, 2011.]
Al contrario, la sintesi degli sgRNA, è un processo discontinuo, ossia avviene “a salti”, come illustrato nella Figura 3. Ogni sgRNA ha, all’estremità 5′, una sequenza di 70 nt chiamata Leader (L), che è presente una volta sola all’estremità 5′ dell’intero gRNA. La sintesi discontinua degli sgRNA dipende da sequenze, chiamate sequenze regolatrici della trascrizione (TRS), presenti a valle del Leader (TRS-L) e che precedono ogni sgRNA (TRS-B) (Figura 1). Le TRS contengono delle sequenze conservate (CS, Conserved Sequences) di 6–7 nt, identiche per il TRS-L e per ogni TRS-B e che permettono l’appaiamento della sequenza CS-L con la sequenza complementare CS-B (cCS-B, complimentary CS-B) dell’RNA intermedio negativo nascente, quando l’RNA si piega per allineare la TRS-L con ogni TRS-B3,4 (Figura 3A).
Questo processo assicura la produzione di ogni sgRNA, che è, sostanzialmente, una fusione tra la sequenza Leader all’estremità 5′ dell’RNA virale e la sequenza dell’sgRNA, preceduto dalla TRS-B. L’intero processo richiede interazioni a distanza RNA-RNA mediate da complessi RNA-proteina. Un esempio di questo meccanismo è mostrato nella Figura 3B.
Figure 3 Sintesi discontinua dell’RNA di SARS-CoV-2
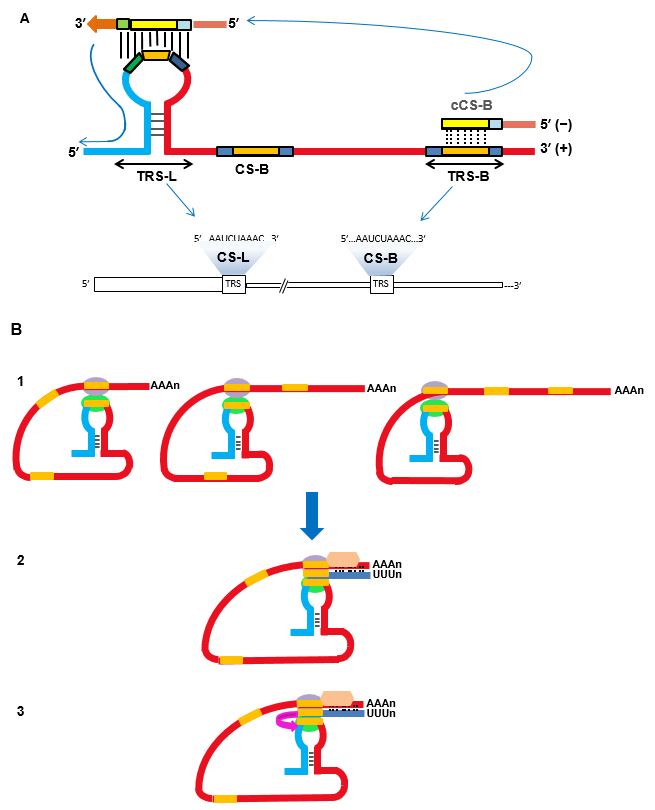
A La sintesi discontinua inizia a ogni TRS-B degli sgRNA. I TRS-B contengono una piccola sequenza identica a quella del TRS-L (sequenza complementare o CS, Complimentary Sequence). Questo assicura lo spostamento (vedi anche B) e l’appaiamento della sequenza complementare alla CS-B (cCS-B) con la CS-L; in questo modo si producono filamenti negativi per ogni sgRNA a partire dalla sequenza leader (L). B Un modello a tre passaggi per la sintesi discontinua. 1. Alcune proteine, rappresentate come sfere, legano la CS (in arancio) della TRS-L e della TRS-B, avvicinando TRS-L e TRS-B. 2. Una volta che è stata prodotta una TRS-B negativa (rettangolo in blu), dalla RdRp (esagono rosa), questa si sposta alla TRS-L, aggiungendo una copia negativa della stessa TRS-L e completando il filamento negativo dell’ sgRNA, che serve da stampo per quello positivo, codificante. [Basato su: Sola et al., Ann Rev Virol, 2015]
Guarda anche il video (in inglese) sulla sintesi continua e discontinua dell’RNA nei coronavirus realizzato nel 2015 da Isabel Sola, Fernando Almazán, Zúñiga e Luis Enjuane per l’Annual Review of Virology:
I salti dell’RNA polimerasi dipendente dall’RNA
La sintesi dell’RNA di CoV-2 è, sostanzialmente, identica a quella degli altri coronavirus umani. Tuttavia, Le caratteristiche di alcuni dei suoi sgRNA suggeriscono meccanismi di sintesi alternativi.
Nella cellula infetta sono sintetizzate, nel complesso, più molecole di mRNA del CoV-2 che della cellula stessa, come se, in qualche modo, l’espressione dei geni cellulari venisse spenta (Figura 4)6.
Il 92% degli sgRNA di CoV-2 è prodotto secondo il meccanismo descritto nel paragrafo precedente e questi sono principalmente rappresentati dagli mRNA per N, S, M, E, 3a, 6, 7a, 7b e 8 (Figura 5A), ma un certo numero di geni, a funzione ignota, hanno una struttura insolita (Figura 5B).
La presenza di alcune sequenze di 3–4 nt, identiche all’estremità 5′ e 3′ di questi geni, potrebbe far ipotizzare un meccanismo di “salto dell’RNA polmerasi” (polymerase jumping), già descritto in altri sistemi (Figura 6).
Figura 4 Trascrittoma in una cellula infetta dal SARS-CoV.2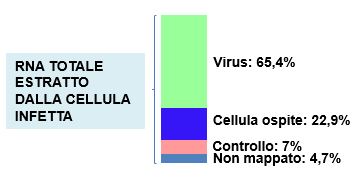
Il 65,4% degli RNA all’interno delle cellule infette appartiene al virus. [Basato su: Kim et al., Cell, 2020.]
Figura 5 Prodotti dell’RNA di SARS-CoV-2

Gli sgRNA canonici (A) e insoliti (B). [Basato su: Kim et al., Cell, 2020.]
Figura 6 Un esempio di “salto della polimerasi”
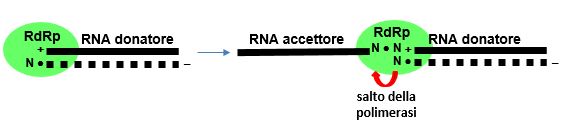
L’RdRp salta dall’estremità 3′ di un RNA donatore a filamento negative all’estremità 3′ di un RNA accettore, appaiando piccole sequenze complementari. [Basato su: Kim et al., Cell, 2020]
Questo processo produce un certo numero di sgRNA a funzione ignota nel ciclo vitale del virus, ma che possono essere potenzialmente tradotti in proteine agli stessi livelli delle proteine accessorie. Sarà interessante verificare se queste proteine effettivamente esistano e che ruolo possano avere.
Ma le cose sono più complicate di così: le modifiche dell’RNA
Le modifiche epigenetiche più note sono quelle a carico del DNA e degli istoni associati, che nell’insieme formano la cromatina, quindi i cromosomi, all’interno della cellula. Anche l’RNA può essere modificato e, in effetti, sono già state osservate modifiche dell’RNA virale in altri virus. È stata osservata l’aggiunta di gruppi metilici (su A o C), la perdita di gruppi amminici dalle basi e l’aggiunta di nucleotidi (per esempio di uracile). Sono state identificate modifiche dell’RNA nel CoV-2 (Figura 7), ma la loro natura non è stata ancora identificata, né tantomeno è stato chiarito il loro ruolo. Tuttavia, si pensa possano essere coinvolte nel controllo della stabilità dei vari sgRNA o che potrebbero contribuire a eludere il sistema immunitario dell’ospite.
Figura 7 Modifiche dell’RNA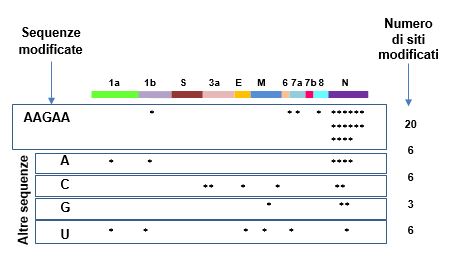
Gli asterischi corrispondono alla posizione approssimativa delle modifiche sul gRNA. Il numero di modifiche è mostrato a destra, il valore più alto si osserva in corrispondenza delle sequenze AAGAA. [Basato su: Kim et al., Cell, 2020.]
Conclusioni
Le caratteristiche biologiche del CoV-2 sono, in larga parte, sovrapponibili a quelle degli altri CoV. Nonostante ciò, il meccanismo di sintesi del suo RNA è molto complesso e, in alcuni casi, sfugge ai meccanismi canonici. In particolare, l’alta frequenza di geni con struttura molto diversa da quelli prodotti dal meccanismo di trascrizione canonico (Figura 3B) potrebbe produrre varianti virali, un punto che merita particolare attenzione se pensiamo ai meccanismi di farmaco-resistenza ed elusione del sistema immunitario che CoV-2 potrebbe evolvere per sopravvivere e propagarsi. Le modificazioni dell’RNA potrebbero, anch’esse, svolgere un ruolo in questi ambiti. Dallo studio in tessuti animali, dove è attiva una risposta immunitaria, si potranno probabilmente ricavare più informazioni su queste caratteristiche. Inoltre, anche lo studio dell’eventuale presenza di modifiche simili dell’RNA in altri coronavirus potrebbero fornire informazioni interessanti per comprendere al meglio le proprietà del CoV-2.
“Per i non esperti”
Che cos’è l’RNA
Il DNA ed l’RNA sono costituiti da una sequenza di “mattoni molecolari” chiamati nucleotidi (nt). Sia nel DNA sia nell’RNA, l’informazione genetica è contenuta in una sequenza di molecole indicate con “lettere”, chiamate basi azotate, che sono uno dei componenti dei nucleotidi, e che per l’RNA sono: adenina (A), guanina (G), citosina (C) e uracile (U). Combinazioni differenti di queste quattro lettere, più o meno lunghe, costituiscono istruzioni diverse per la sintesi di tutti i prodotti i proteici. Ciò è vero sia per gli organismi superiori sia per i microrganismi, come i batteri, nonché per i virus.
Mentre nelle cellule il DNA è sempre la molecola in cui è depositata l’informazione genetica che può essere ereditata, e l’RNA svolge altre funzioni, nei virus questa funzione può essere svolta sia dal DNA (virus a DNA) sia dall’RNA (virus a RNA).
Nelle cellule l’RNA svolge funzioni diverse e può assumere strutture differenti. Tra le varie funzioni, concorre alla produzione delle proteine mediante tre tipi di RNA:
- l’RNA messaggero (mRNA), che copia l’informazione contenuta nel DNA e la porta nel citoplasma);
- l’RNA transfer (tRNA) che permette la traduzione del codice dell’RNA in quello delle proteine;
- l’RNA ribosomiale (rRNA), che costituisce la struttura all’interno della quale sono sintetizzate le proteine.
l processo che porta alla sintesi delle proteine è chiamata traduzione perché implica una fase di traduzione del codice genetico dell’RNA in quello delle proteine.
Le RNA polimerasi
Le RNA polimerasi sono le proteine che sintetizzano le molecole di RNA unendo tra loro i singoli nucleotidi. Ne esistono di diverso tipo. Quelle che uniscono i nucleotidi copiando la sequenza da un altro filamento di RNA sono dette RNA polimerasi dipendenti dall’RNA.
L’epigenetica
L’epigenetica è la branca della scienza che studia le risposte del genoma agli stimoli intracellulari, ambientali, meccanici ecc. Le modifiche epigenetiche sono i “marchi” di queste risposte e, in sostanza, non sono altro che modifiche chimiche che possono essere apportate ai genomi e anche alle proteine. Le cosiddette “macchine epigenetiche” sono responsabili di queste modifiche e sono complessi multiproteici attivati dagli stimoli sopra menzionati. Le modifiche epigenetiche cambiano le caratteristiche di un genoma, per esempio dando istruzioni su quali geni devono essere accesi o spenti, in modo che la cellula possa rapidamente rispondere a segnali interni o esterni.
Bibliografia
- Lin, J. T. et al. Safety and immunogenicity from a phase I trial of inactivated severe acute respiratory syndrome coronavirus vaccine. Antivir Ther 12, 1107-1113 (2007).
- Yong, C. Y., Ong, H. K., Yeap, S. K., Ho, K. L. & Tan, W. S. Recent Advances in the Vaccine Development Against Middle East Respiratory Syndrome-Coronavirus. Front Microbiol 10, 1781, doi:10.3389/fmicb.2019.01781 (2019).
- Snijder, E. J., Decroly, E. & Ziebuhr, J. The Nonstructural Proteins Directing Coronavirus RNA Synthesis and Processing. Adv Virus Res 96, 59-126, doi:10.1016/bs.aivir.2016.08.008 (2016).
- Sola, I., Almazan, F., Zuniga, S. & Enjuanes, L. Continuous and Discontinuous RNA Synthesis in Coronaviruses. Annu Rev Virol 2, 265-288, doi:10.1146/annurev-virology-100114-055218 (2015).
- Masters, P. S. The molecular biology of coronaviruses. Adv Virus Res 66, 193-292, doi:10.1016/S0065-3527(06)66005-3 (2006).
- Kim, D. et al. The Architecture of SARS-CoV-2 Transcriptome. Cell 181, 914-921 e910, doi:10.1016/j.cell.2020.04.011 (2020).


 Scarica il PDF dell'articolo
Scarica il PDF dell'articolo
Enrico Lantero
Grazie per le utilissime info. Segnalo un errore nel riquadro “Per i non esperti” … nella frase “nei virus questa funzione può essere svolta sia dal DNA (virus a RNA) …” dovrebbe essere credo scritto “nei virus questa funzione può essere svolta sia dal DNA (virus a DNA)… “. Grazie per l’attenzione.
Redazione Autore articolo
Grazie per la segnalazione, abbiamo corretto.
Claudia
Grazie davvero per queste risorse! Non conoscevo alcuni meccanismi e, di altri, mi mancano dettagli molecolari (es. i salti della polimerasi).
Di certo sono risorse davvero interessanti e fomentano la mia (mai persa) voglia di tornare alla ricerca!
Grazie!
Giulio Ferrari
Buona spiegazione.
Occorre ampliare con le tecniche impiegate per l’individuazione dei processi trascrizionali e l’identificazione delle proteine sintetizzate.
Cordiali saluti